
iGEM

0.1 分子生物学基础知识
螺距3.4nm。Tm与溶液中离子强度、DNA均一性也有关。>90%的非编码序列。一个复制子只含一个复制起点。
转录:封闭复合物→开放复合物→三元复合物。RNAP,含有多个亚单位。转录单元——启动子+转录区域+终止子。识别-起始-延伸-终止。
原核生物启动子共同特点:识别-结合-起始三个位点,常和操纵子相邻。
真核生物启动子RNAP II识别:**-25~-30(TATA区/Hogness区,类似Pribnow)、-70~-78(CAAT区)**。Terminator反向重复互补序列。
增强子常在200bp之外,可以在基因内部——远距离效应、无方向性、顺式调节、无物种与基因特异性、有组织特异性、有相位性(与DNA构象有关)、部分反应外部信号。
常识数据(细菌数据):
转录mRNA速度:2500 nt/min≈14 codon/s;
翻译速度:15 codon/s;
DNA复制速度:800 bp/s。当基因开始表达,约2.5min出现mRNA,再过半分钟可检测到对应蛋白质。
原核mRNA半衰期短,无或仅短的polyA。上游Shine-Dalgarno序列保守区/Ribosome Binding Site与16S rRNA 3’端反向互补。多顺反子,多RBS。
加帽:入胞质、防降解、IF III起始因子识别促翻译。polyA可以是转录产生或后加,入胞质、稳定性、有效循环。U1 snRNA以碱基互补方式识别mRNA 5’端剪接信号。
终止密码子不算做ORF。UAA-Ochre赭石、UAG-Amber琥珀、UGA-Opal乳白。
Trp仅1种(UGG)、9种aa有2个codon、1种有3个、5种有4个、3种有6个。N端Met在多肽链合成完之前就会被N氨肽酶切除。Ser是最广泛的磷酸化位点。
分子伴侣:帮助其他多肽正确折叠、组装、运转、降解。NES:Leu&Ile等疏水性aa富集。被CRM1识别并被携带出核。
0.2 生物技术基础知识
克隆的基因不一定能表达、克隆载体不一定是好的表达载体。
表达载体的要求:控制转录、控制翻译、控制表达蛋白稳定性、有利于蛋白纯化过程(分泌获取)。
启动子终止子序列特点、RBS结合强弱、翻译效率、拷贝数、稳定性、转入基因定位(质粒or染色体)、表达蛋白质定位。
pET系列载体:【T7噬菌体基因10启动子+目的基因】导入到大肠杆菌染色体上,lac启动子+T7 RNAP。IPTG诱导产生T7 RNAP,进而产生目的蛋白。有1h以上的延迟。
任何在某种生物体中使用不足5%~10%的密码子都会因为对应tRNA稀少而导致蛋白产量降低。特别是两个及以上的稀有密码子靠近mRNA 5’端时对蛋白质表达影响最大。
The reasons behind Metabolic Load:能量需求、溶解氧不足、过度表达的蛋白的运输回堵塞其他重要蛋白的运输通道而影响其细胞定位与功能、外源蛋白有时可能把重要必需的中间产物变成无关且甚至有毒的产物。
Influence:降低细胞生长速率、丢失部分重组质粒或质粒结构的一部分,固氮与蛋白质合成受影响,胁迫反应导致蛋白降解、翻译错误增加(校正需要GTP。在治疗性药物中的体现就是特异性与稳定性发生变化而诱发免疫反应)。细胞缺氧时菌体会迅速进入静止期,并大量产生蛋白酶。
Solutions:蛋白酶缺陷株、使用专性需氧菌、表达氧结合类蛋白质解决氧气溶解问题(血红蛋白)。
哺乳动物细胞缺点:长得慢、需要更加严苛的生长条件、会感染动物病毒。优点主要是翻译后修饰(“原汤化原食……”)。
常用哺乳动物的表达载体特点见下。把内含子插入到启动子和多克隆位点之间,往往可以提高表达量。翻译起始在Kozak序列,最好也有5’/3’-UTR,为了提高翻译效率和mRNA稳定性,一般用的是人β-globin的。
- 基于猿病毒40(SV40)DNA:装载外源基因空间有限。仍含有复制所需的早期基因和病毒粒子生产所需晚期基因。
- 腺病毒:装载大。
- 牛乳头瘤病毒:多拷贝。
- 腺联病毒载体:可以整合到染色体上特意位点。
1. 合成生物学概论
traditional biology: deconstruction; synthetic biology: re-synthesis.
Nowadays Limits: limited in a few toy-systems; may solve “created” problems
meaning: apply the basic principle of biology, rationally design and build new (man-made) life systems that can perform a predetermined function. 可设计性、人工可调、定量化、模块化。
goal: to make the engineering application of molecular biology systematic, quantitative and simple.
2000年后,基因组学、生信、系统生物学的发展开启了合成生物学时代——基于系统生物学的遗传工程。基因线路、人工染色体&细胞器、细胞与多细胞体系、人工生物系统的定量表征&模拟。
Synthetic Repressilator:生物振荡系统。
Collective synchronization: 基于细胞通讯的同步化振荡,群体振荡。
Toggle switch: 双稳开关(t触发、溶原裂解转化)。
感受内外源信号→信号处理→决策制定→多输入输出→动态环境适应。
生命组织特征:
- chemical complexity and microscopic organization. 非物理隔离的分子组织形式。
- extracting, transforming and using energy from the environment.
- sense and respond. 随时空变化的复杂力学行为。
- change over time by gradual evolution.
- self-replication and self-assembly
- different organisms’ components and regulated interactions.
具有适应性的网络都可以被分为两大类:负反馈与不一致的前馈网络。
RBS计算器的生物物理模型;Cello细胞编程软件;Metabolic Engineering。有机小分子、生物大分子、生物分子网络设计、细胞设计。
2. 合成生物学挑战与工程化标准
No reliability and predictability. Cost. Extremely high complexity。
复杂度的瓶颈,反摩尔定律(越来越难发明新药)。从复杂的天然生物系统中发掘元器件,进行模块化与标准化,之后元器件的再组装,进行适配于微生物细胞的元件精细加工。
基因元件(生物功能的基础单元)→根据工程原则设计DNA序列→全幅DNA合成与组装技术→导入底盘细胞发挥功能。
基因元件的设计原则:简化原则/模块化、正交性好/绝缘化、无or低细胞毒性。
工程化思想:标准化、解耦合(decoupling)、可抽提化(abstraction)、可进化性、自繁殖性。
3. 合成生物学元器件
What is biological part?——DNA fragments with basic biological functions、not encode multiple functions、compatible(即插即用)、work robustly(稳定/鲁棒)。
Entity of “parts”: transcription&translation、post-translational regulators、signal processing、metabolic enzymes、actuators.
repressors example: LacI、lambda CI;AraC;CytR.
T7 RNAP: 单体聚合酶。识别特异性启动子。
Tet-On system, Riboswitch sensor(结构可以是多种多样的。不同检测物通过SELEX构建。不同核糖开关关联起来可以构成逻辑门), Toehold switch(一个trigger结合mRNA 5’端toehold打开茎让RBS暴露。Application - virus test), Quorum sensing(细菌群体感应,蛋白靶向小分子), Protein tags(Ubiquitin + DD domain + SSA-tag + PEST + NLS + NES + Degron + CAAX box + XFP + FLAG + HV + V6 + SNAP), Integrase/Recombinase(整合酶/重组酶,保证酶与识别位点的正交性。做one-bit memory circuit).
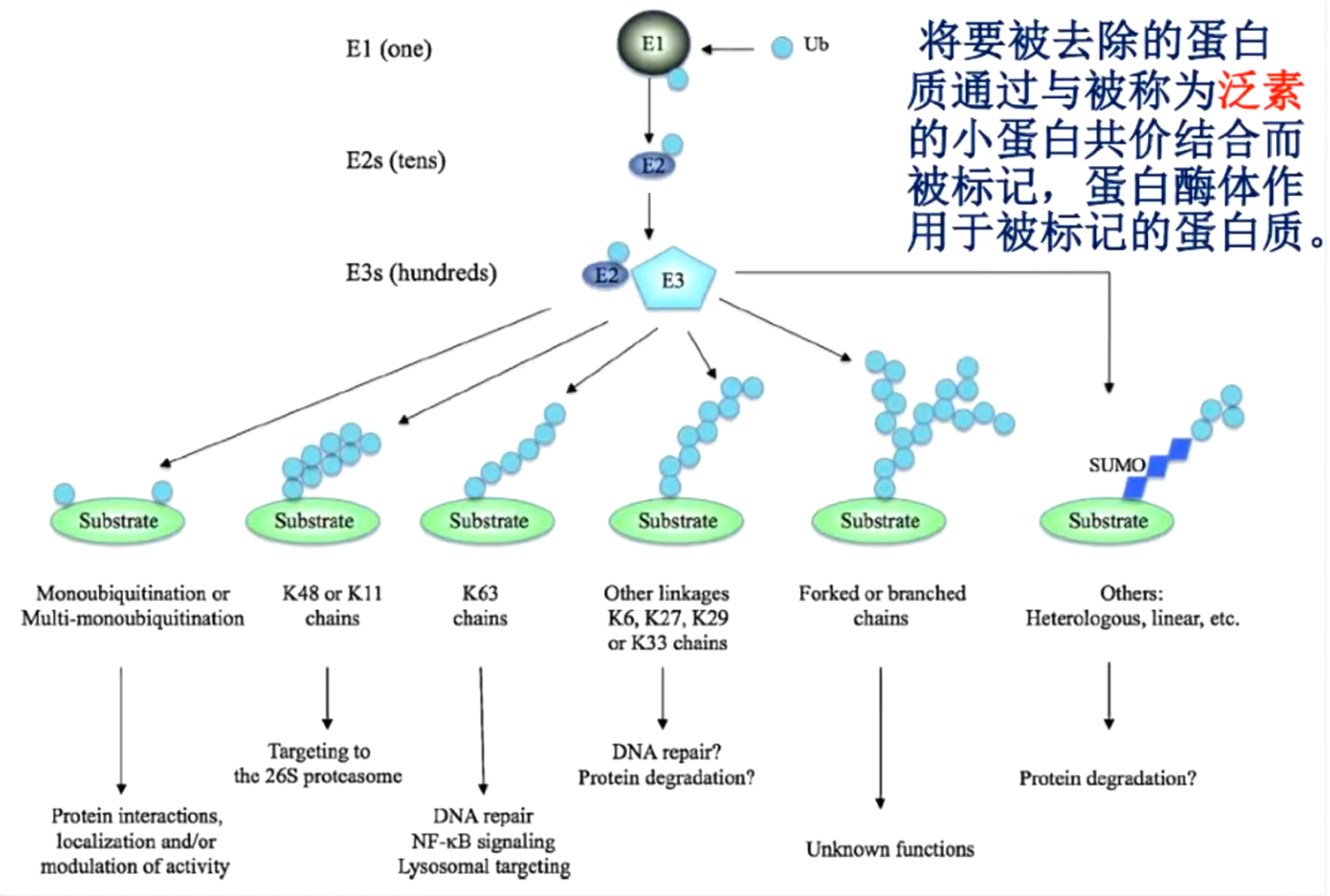
元件抽象化标准化库:“Biobricks”。Eg: pSB【质粒骨架标识】**#【复制起点】X【抗性标记】#**【版本号】。
4. 蛋白质元件与设计
纤维蛋白、球蛋白、膜蛋白(Trp&Tyr: membrane interface anchors. Positive inside rule)。结构层次:残基序列→二级结构→超二级结构→结构域与三级结构→多亚基结构与分子聚合体。跨膜约20aa。功能举例:受体、配体、酶、支架蛋白、 基因调控蛋白、蛋白质标签、功能效应蛋白。
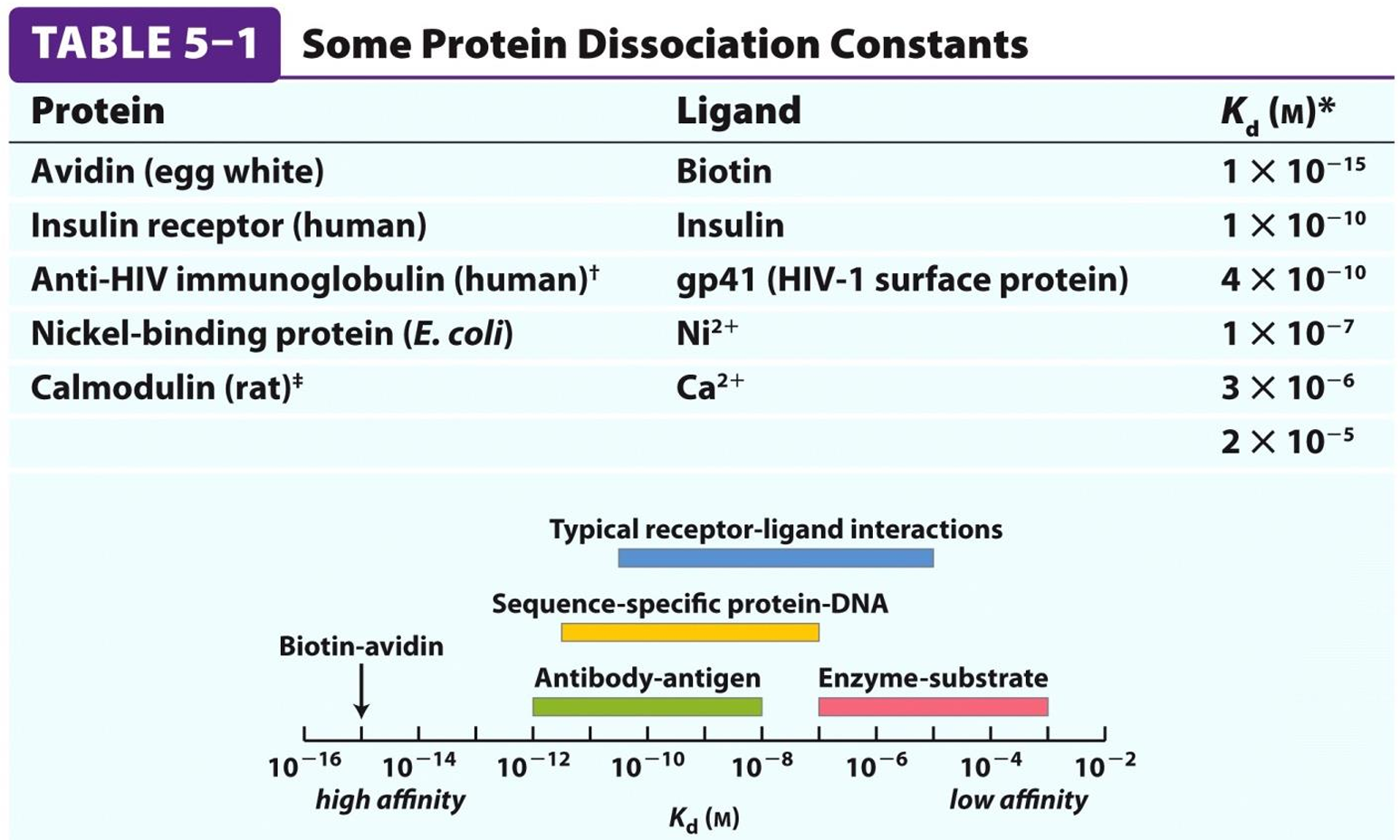
解离常数:越小越强。
蛋白质设计思路:突变、定向进化、de novo design从头设计、”模块化”结构域重组。锌指蛋白是第一个利用程序辅助设计的全新蛋白实例。
Challenges: computational and experimental demanding, low accuracy, conformational changes upon binding(构象变化).
Synthetic protein scaffolds provide modular control over metabolic flux.
5. DNA组装技术/分子克隆
独立于染色体之外的DNA。哺乳动物细胞中极少数质粒可以复制。
promoter、selectable marker、antibiotic resistance gene、origin of replication.
退火温度根据引物与模板的结合能选择。根据酶的合成速度来判定延伸时间,延伸太久有可能会有非特异性位点产生。
限制性内切酶:sticky end and blunt end. 同尾酶(isocaudamer): 识别位点不同,切出粘性末端相同。
感受态transformation:**低温氯化钙(进入感受态)+42℃×2min(退出感受态)**。
基因工程的第一个成功案例1983-human insulin from recombinant DNA technology
Biobrick:常用同尾酶方法。是符合是符合限制性内切酶组装标准的DNA序列。这些构建块用于从单个部件和具有定义功能的部件组合中设计和组装更大的合成生物回路,然后将其整合到活细胞中以构建新的生物系统。
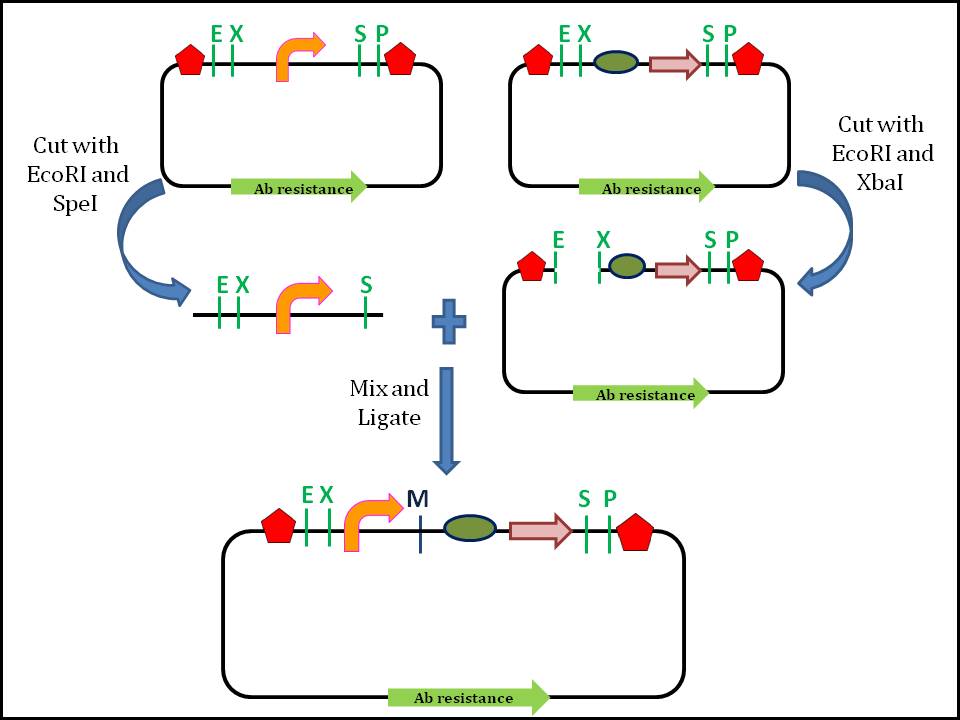
不同同尾酶混合位点形成一个“scar”,因为不能再被切割。如果想形成一个融合蛋白,需要考虑scar被翻译成氨基酸对蛋白功能可能造成的影响。而如果是引入柔性氨基酸(Gly&Ser),则对融合蛋白的两个部分影响较小。
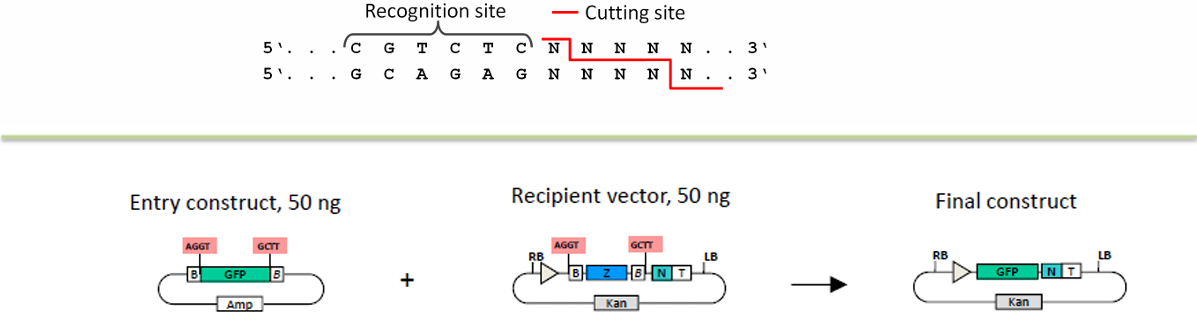
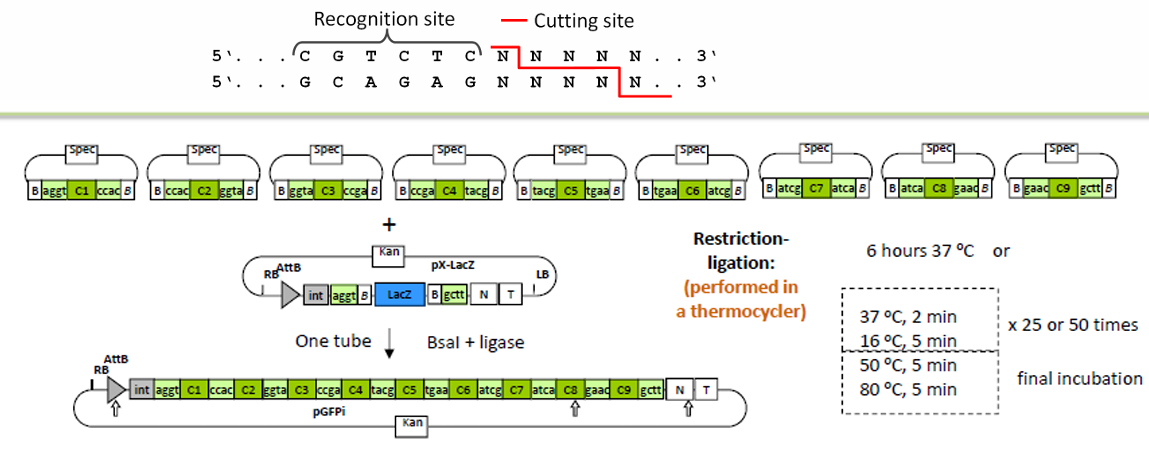
Golden Gates:利用II类限制性内切酶,识别序列在切割位点之外。
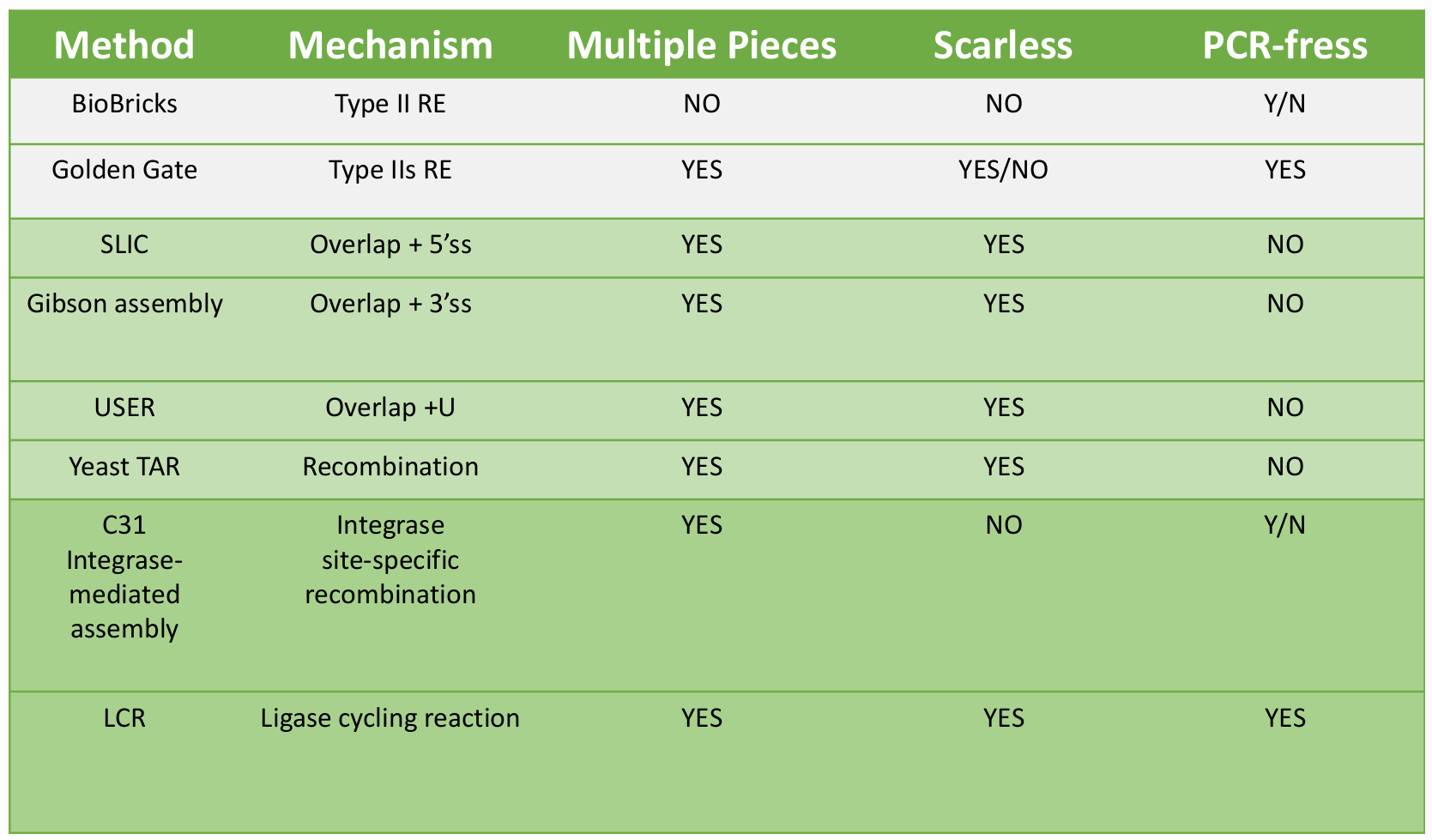
不基于限制性内切酶的方法:circular polymerase extension cloning (CPEC)、sequence and ligation-independent cloning (SLIC)、Gibson (in vitro)、Yeast TAR (in vivo).
CPEC: 类似于PCR,效率低,最后连接是在菌体内进行。
LIC: T4 DNAP,只加对应dNTP可以让聚合酶停留在对应特异位点,还是靠粘性末端靠在一起。
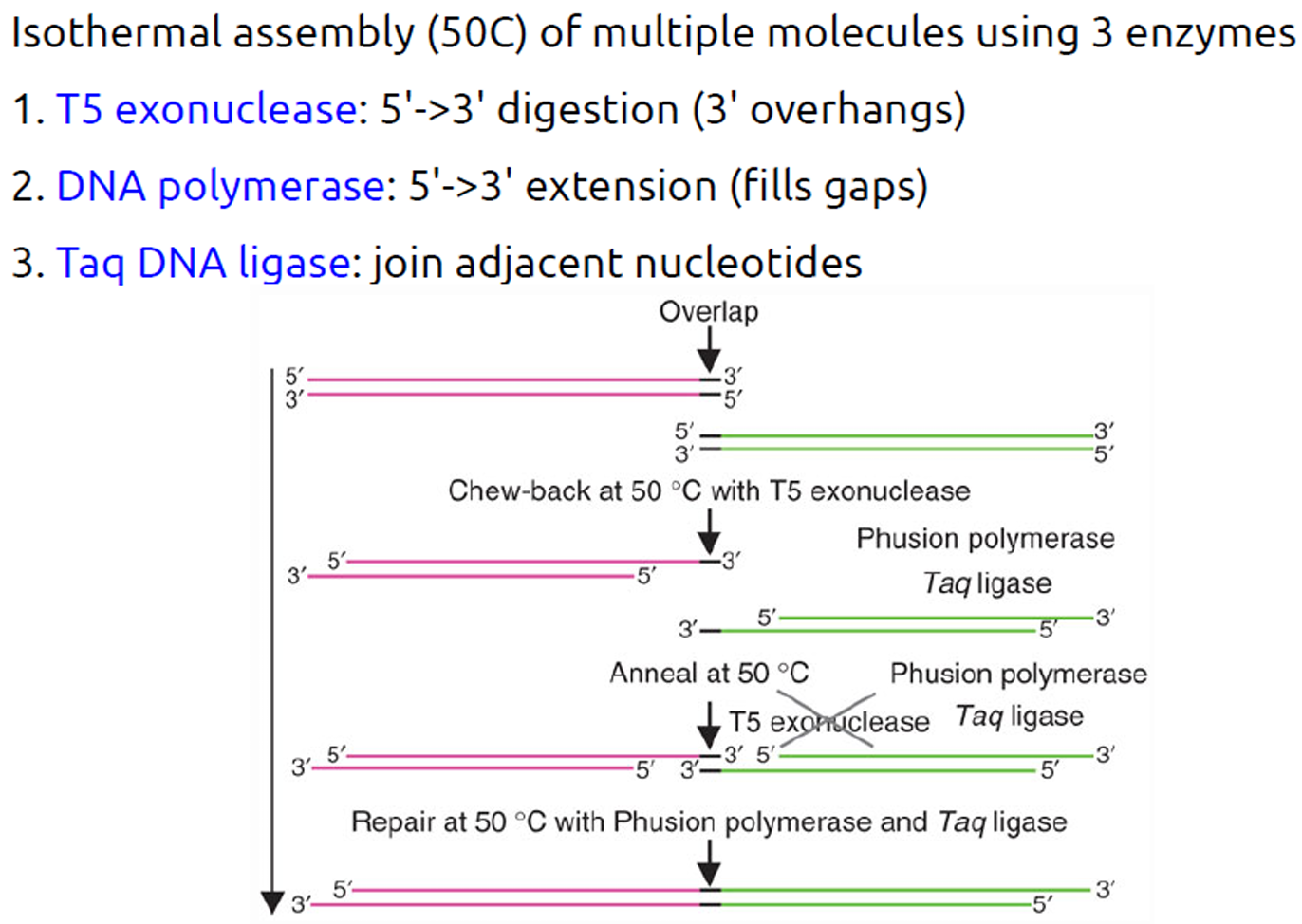
Gibson: 对PCR产生的平末端片段进行外切(T5外切酶),创造粘性末端(末端overlap一小段序列相同),Taq补上缺失部分。一个体系里三个酶即可完成(Phusion DNAP + Taq ligase + T5 exonuclease)。overlap 20~60nt,连接片段30~50kb,大于8个片段轻松carry。
Yeast TAR: 特大片段重组时更常见。直接把大序列片段放到酵母细胞里,让其打工连上。错误率高,那就筛选罢。
图解另外几个不常用非限制性内切酶克隆方法
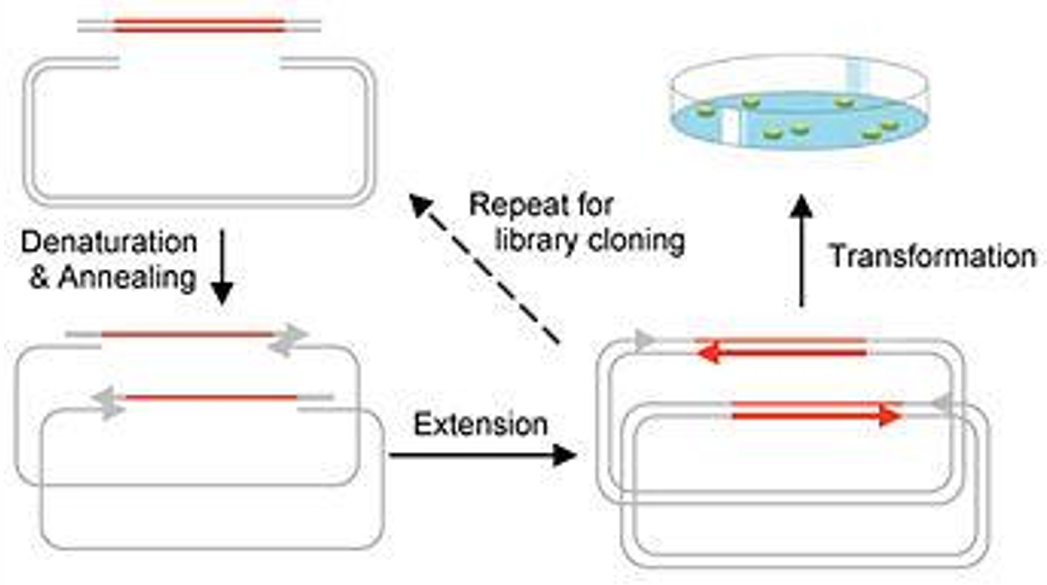
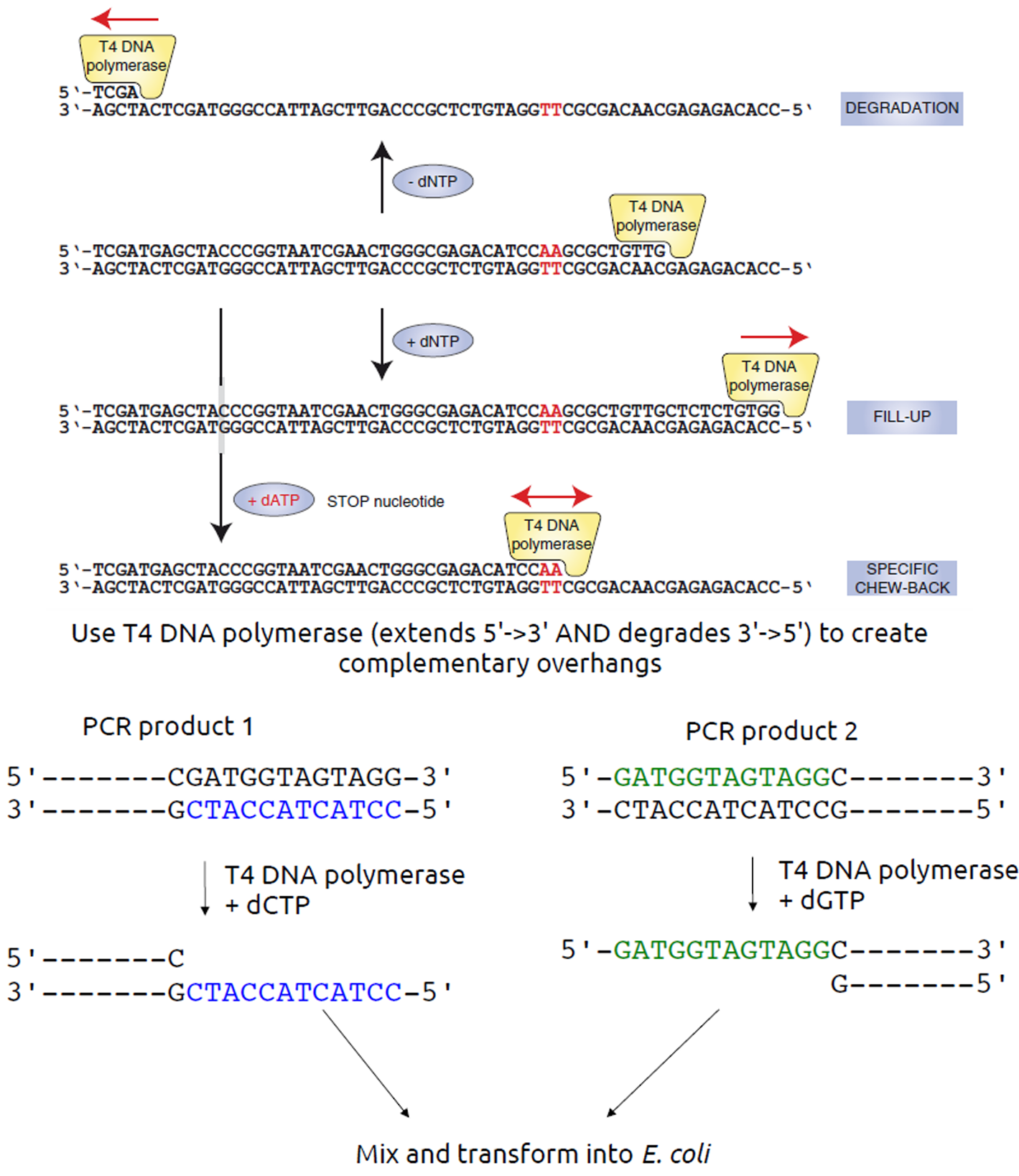
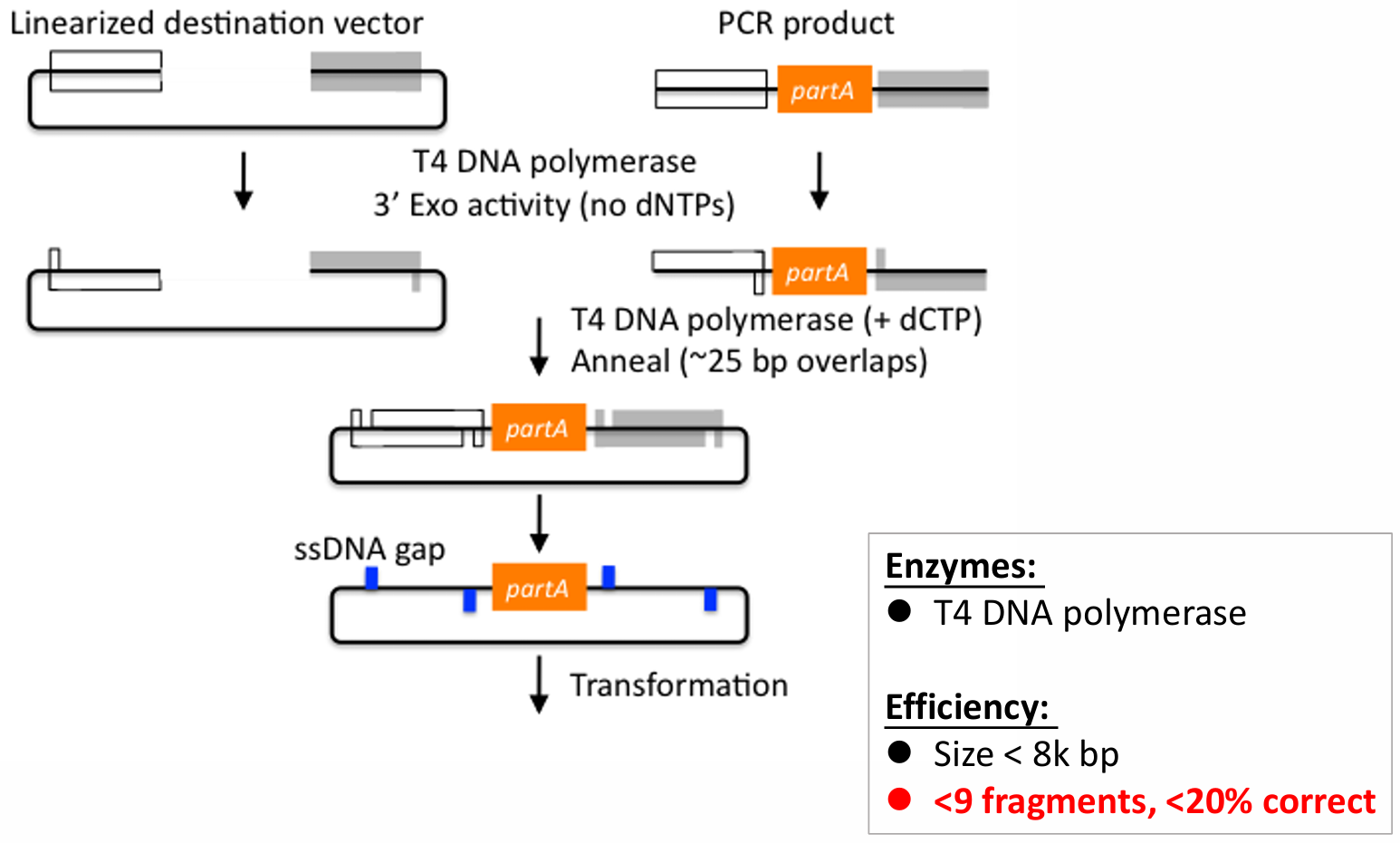
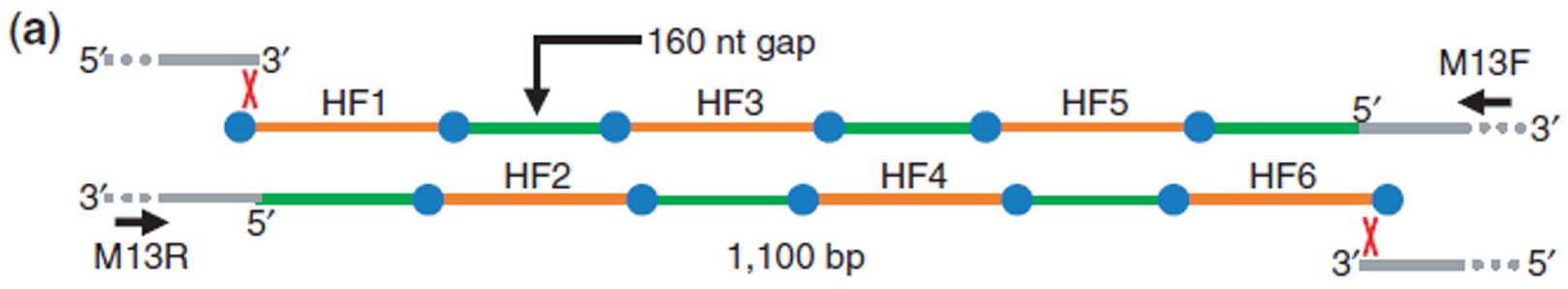
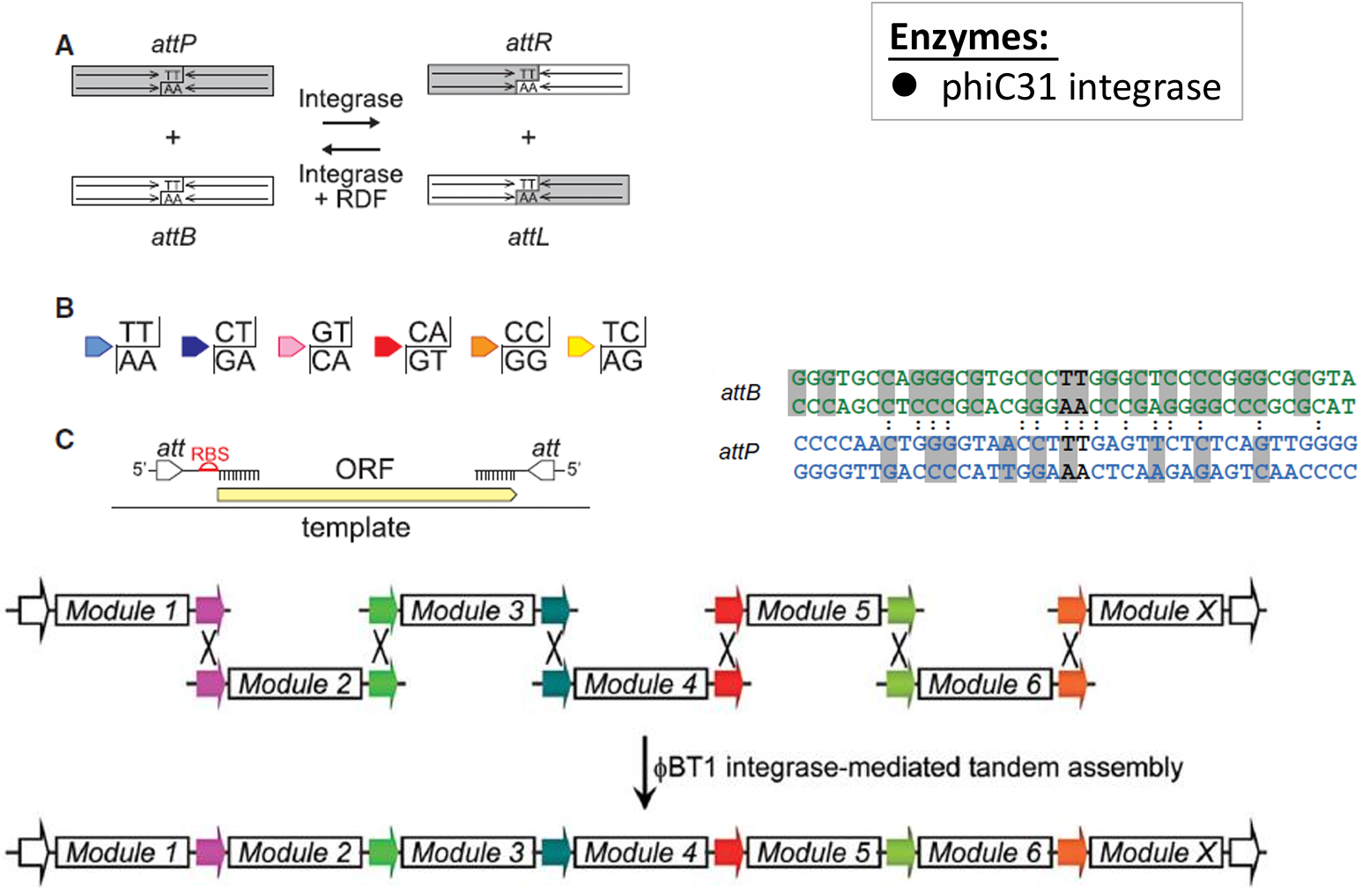
另外还有整合酶方法(in vitro site-specific recombination)、LCR(ligase cycling reaction,自己做个”小胶带“)。
余项补充
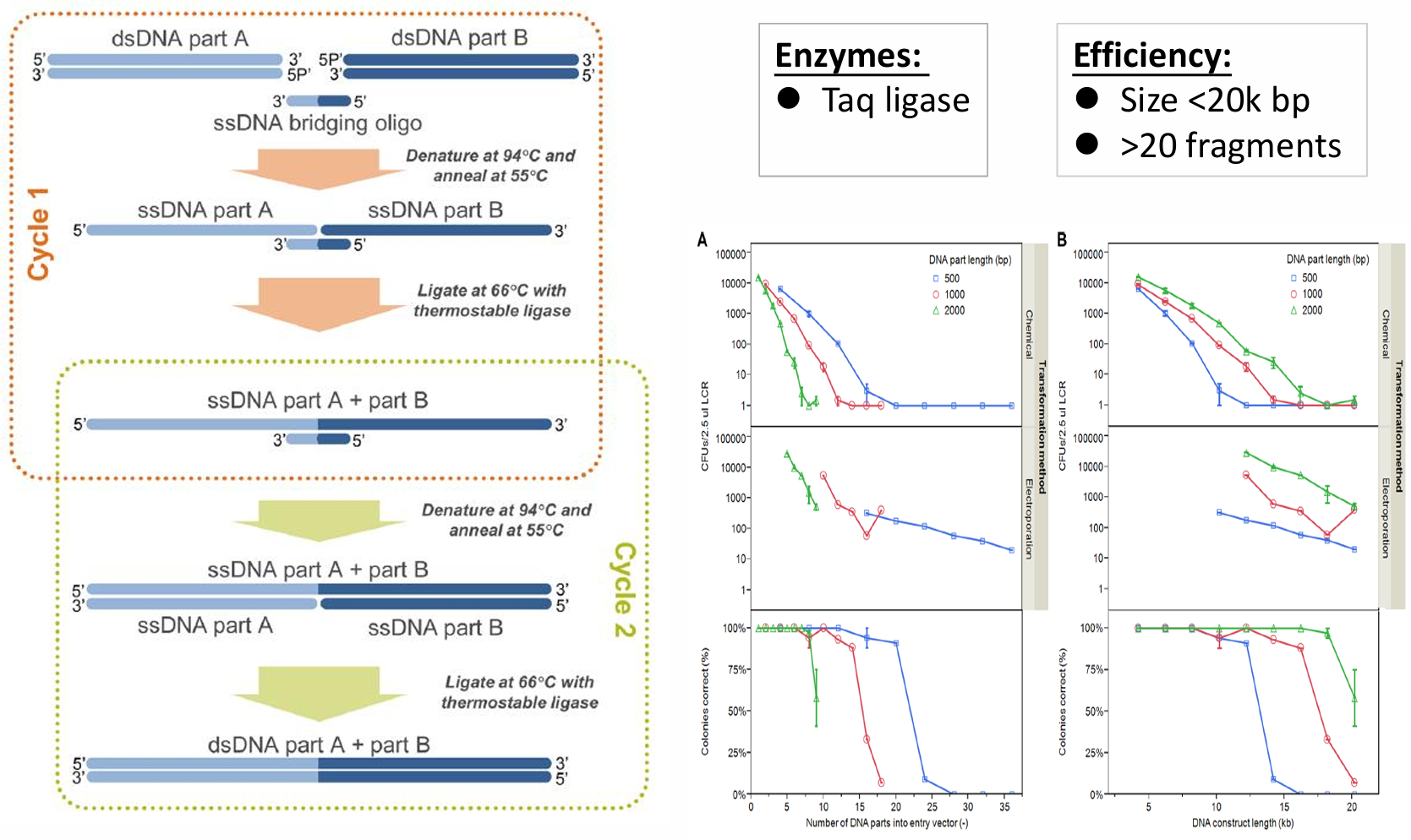
最后是总结:
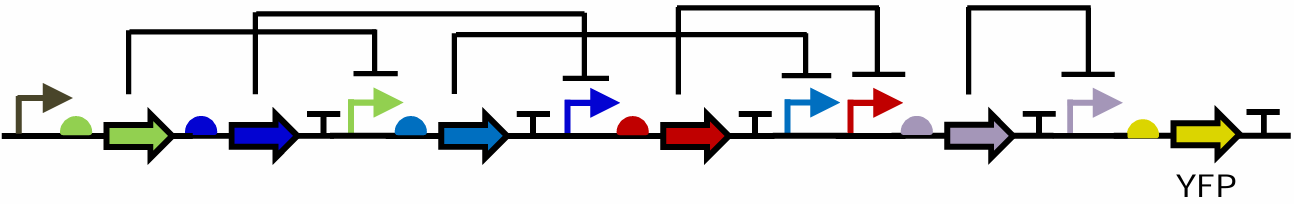
6.1 生物网络设计-基因线路
Logic Gates、Oscillator、Pattern Formation (Turing).
Eg-AND: 单独蓝色和绿色表达都会让灰色输出,最后无YFP,只有蓝色和绿色都未表达才会表达出YFP。(有点像很多红石火把)
真值表,根据输入来得到不同输出的16种逻辑门。小圈是not进行取反。
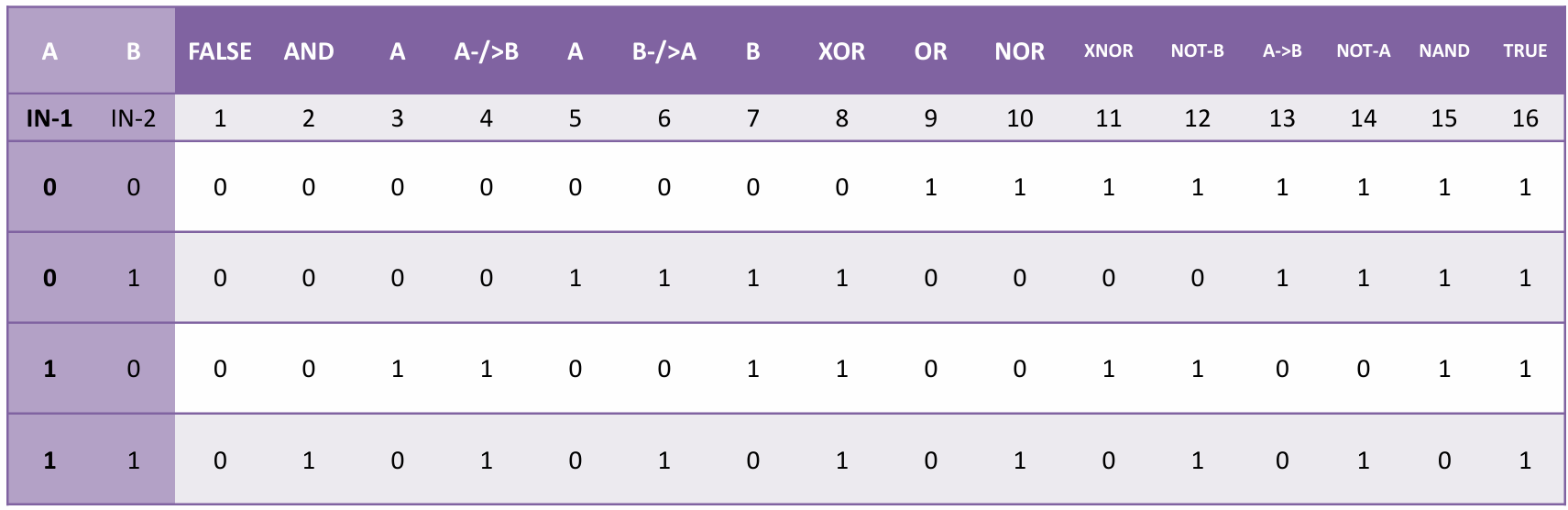
布尔代数的基本算符——AND(与门)、OR(或门)、NOT(非门)、NAND(与非门)、NOR(或非门)、XOR(异或门)、XNOR(同或门)。
| 基本算符 | 记作 | Eg |
|---|---|---|
| AND | + | a+b |
| OR | · | a·b |
| NOT | ‘ | a’ |
| NAND | ()’ | (ab)’=a’+b’ |
| NOR | ()’ | (a+b)’=a’b’ |
| XOR | (+) | a(+)b=a’b+ab’ |
| XNOR | ((+))’ | (a(+)b)’=a’b’+ab |
根据实际情况给出真值表(各开关离散状态枚举)👉将真值表变为布尔代数逻辑表达式👉布尔代数式的电路表示。
实际中最后才是用实际的启动子-终止子-整合酶等等设计出实际的逻辑门基因线路。
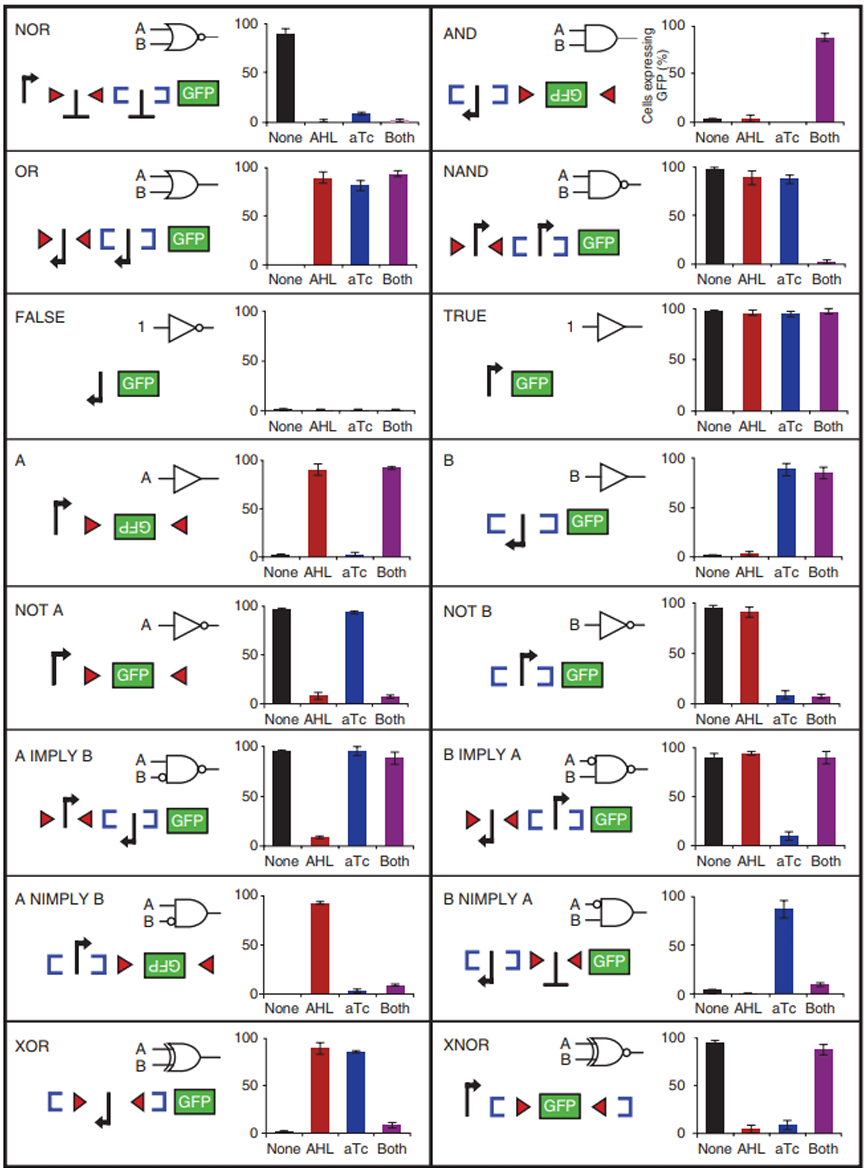
Memory: Bistable switch (双稳开关,参见裂解溶原转换).
Recommendation: Programming a Pavlovian-like conditioning circuit in Escherichia coli.
这里也给出图灵斑图的形成模式:
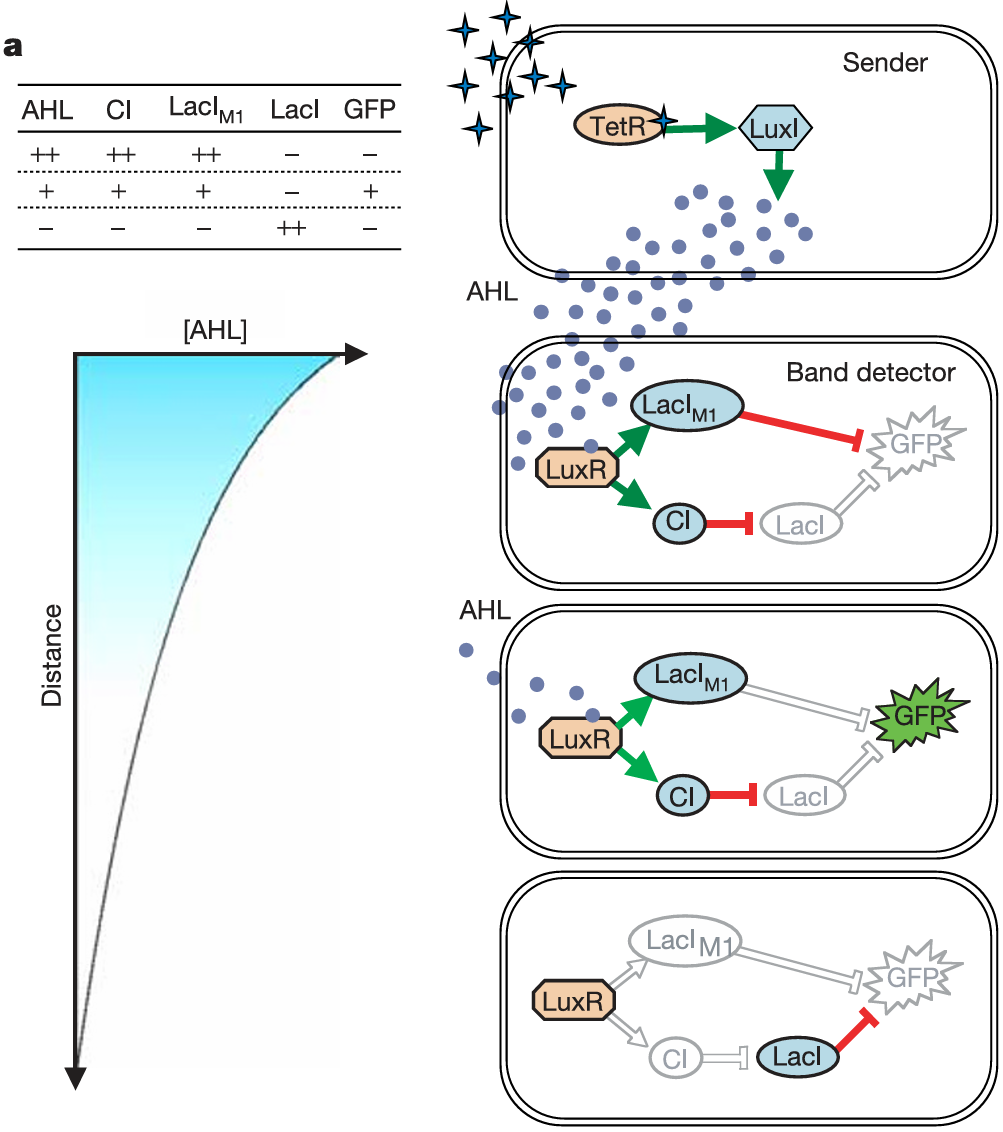
6.2 生物网络设计-信号网络
Toggle switch, Quorum sensing, Oscillator, Engineered Scaffold, Polarization, Synthetic MAPK.
Oscillator: Cell cycle, Circadian clock, Repressilator. 抽象出简化模型。
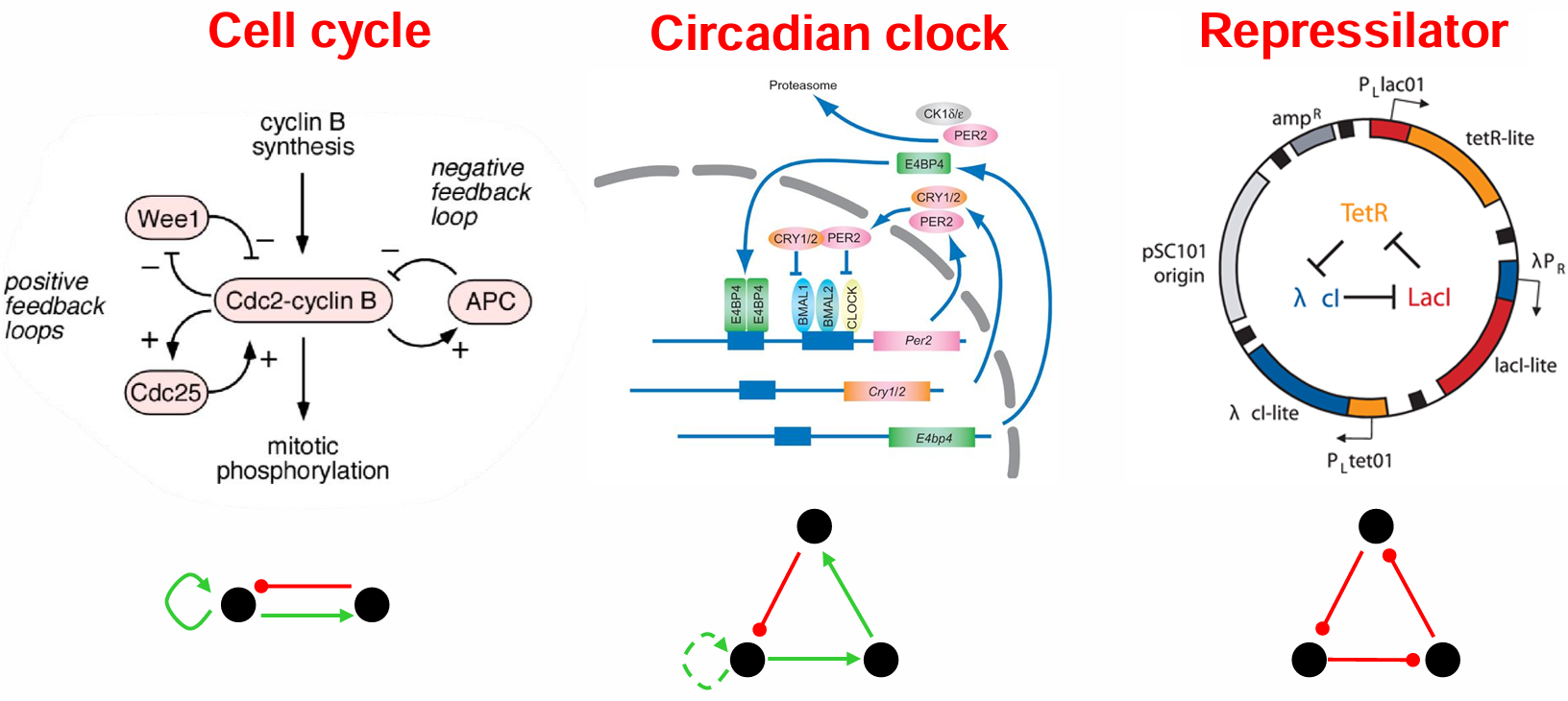
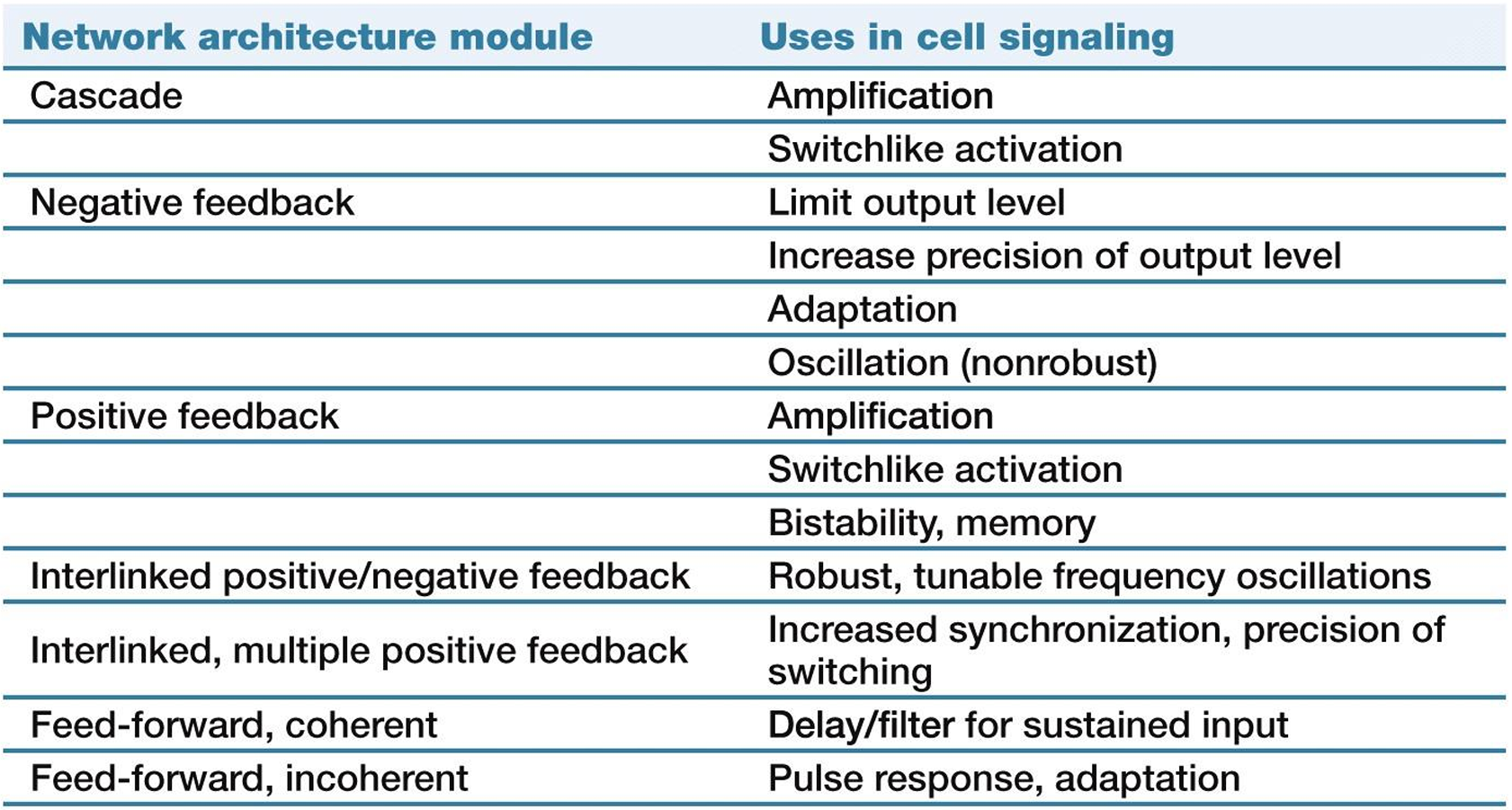
网络工作模型及其应用:
- 正反馈→双稳态+记忆;
- 负反馈→限制输出水平和输出时间+增加输出水平准确性+适应+振荡而不鲁棒;
- 内联正负反馈(interlinked positive/negative feedback)→鲁棒性、可调振荡频率;
- 内联多重正反馈(interlinked, multiple positive feedback)→增强同步化,准确转变;
- 一致前馈(feed-forward, coherent):延迟/筛选稳定输入;
- 不一致前馈(feed-forward, incoherent):暂停反应,适应。
Fan in(多对一)、Fan out(一对多)。
激酶作用:构象改变、互作、降解、定位。
IOQ:ultrasensitive(超敏,有一个input-output急剧上升的阶段)
胞内活动的时间定量:转录报告物-大于15min、定位改变-1min、蛋白互作-小于10s
细胞抑制性:每一个单细胞表现出对应表型有一定随机性,主要是上面的时间定量存在差异。如果观察精度不高,那就会粗略认为整体呈现平均变化。
7. 生物系统建模
常微分动力学方程、随机模拟方法、生物物理模型、布尔逻辑模型、唯象模型(有点形而上的感觉)。
自治系统、非自治系统(演化规则随时间变化,更多一维)、自由度(指相空间的维数)。低维动力学系统如阻尼振子。
Fixed Points and Stability.
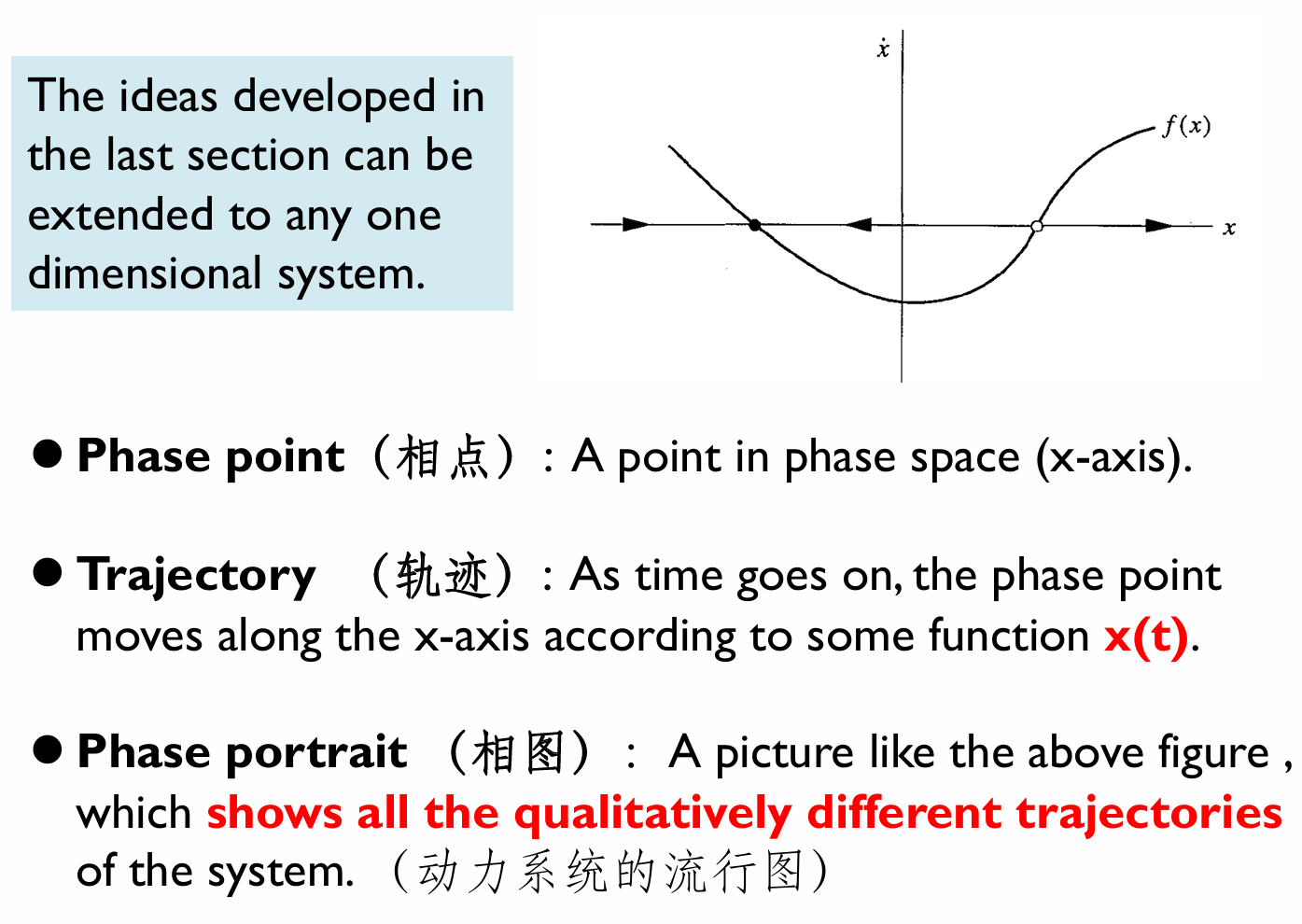
基因转录建模的基础类型,ppt还是讲得比较清楚,仅作思路引导,并没有特别的掌握要求:
搬运于此,想看自戳
基于化学动力学质量作用定律给出的空启动子(D)和结合启动子(C)的动力学方程,打一个点是牛顿写法的一阶导数。



展示了在多种不同效应转录因子结合不同位点时的转录过程建模,需要遍历各种情况,因为在真是细胞中的总转录活性是不同结合情况的宏观体现。至于X、Y是几次幂,这是看启动转录过程中需要多少个相同种转录因子结合,因为究其发源是质量作用定律。

如果是相同位点,那就只有下面的情况。

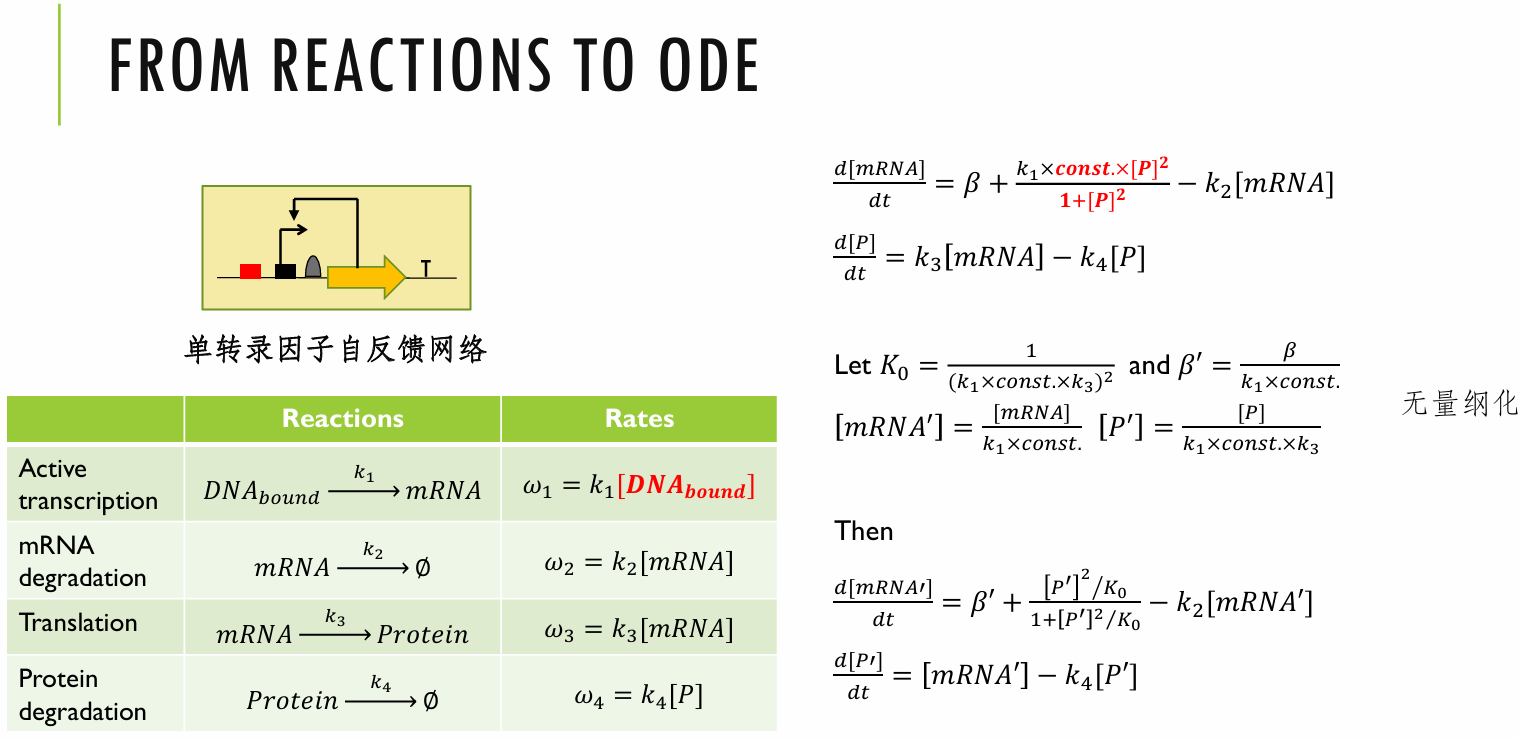
然后是单转录因子自反馈的建模,想看自戳
模型中mRNA的合成受到产物的反馈影响,而蛋白质合成与分解被简单认为是两个一级反应。
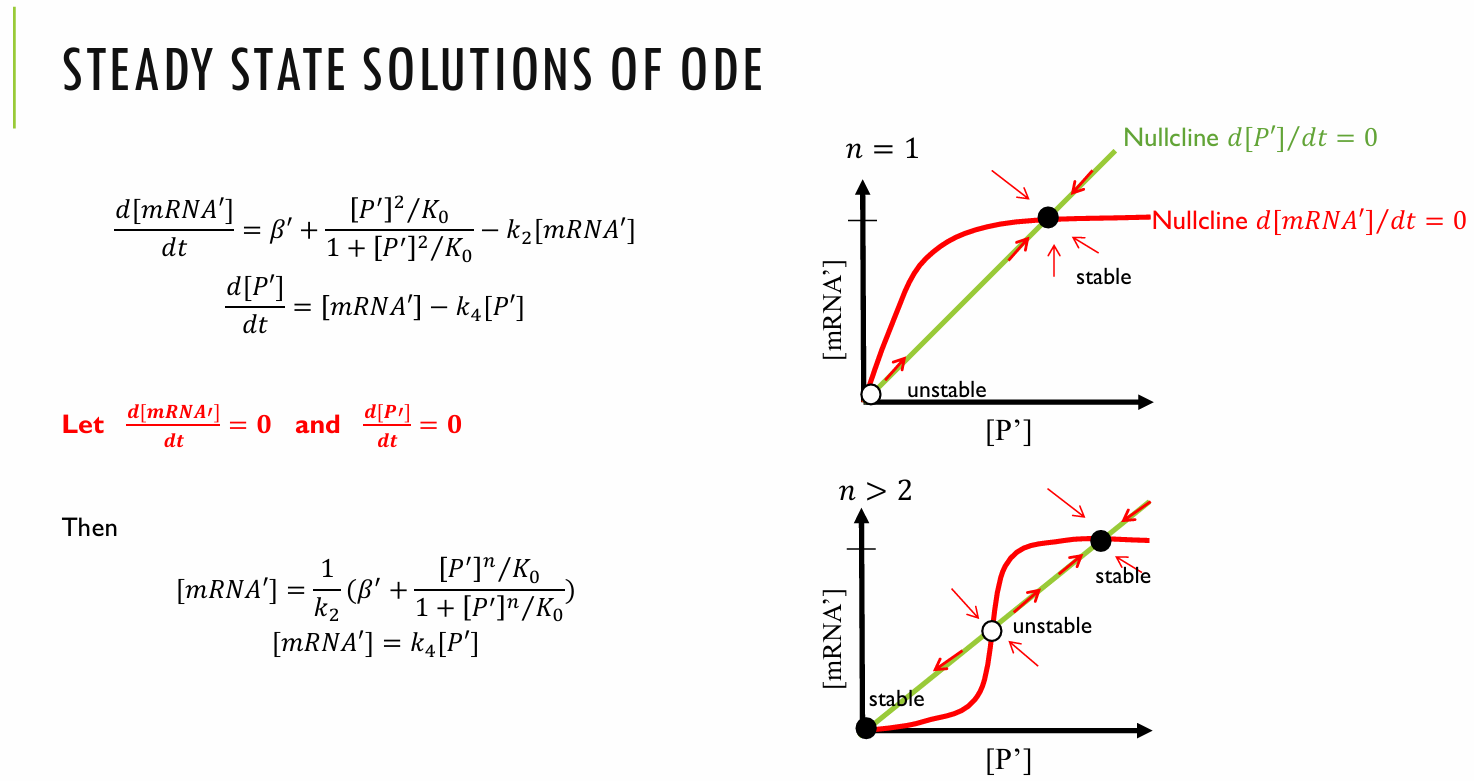
最后产生的蛋白以何种数量方式反馈影响自身转录会影响系统存在几个稳定点,以及稳定与否。
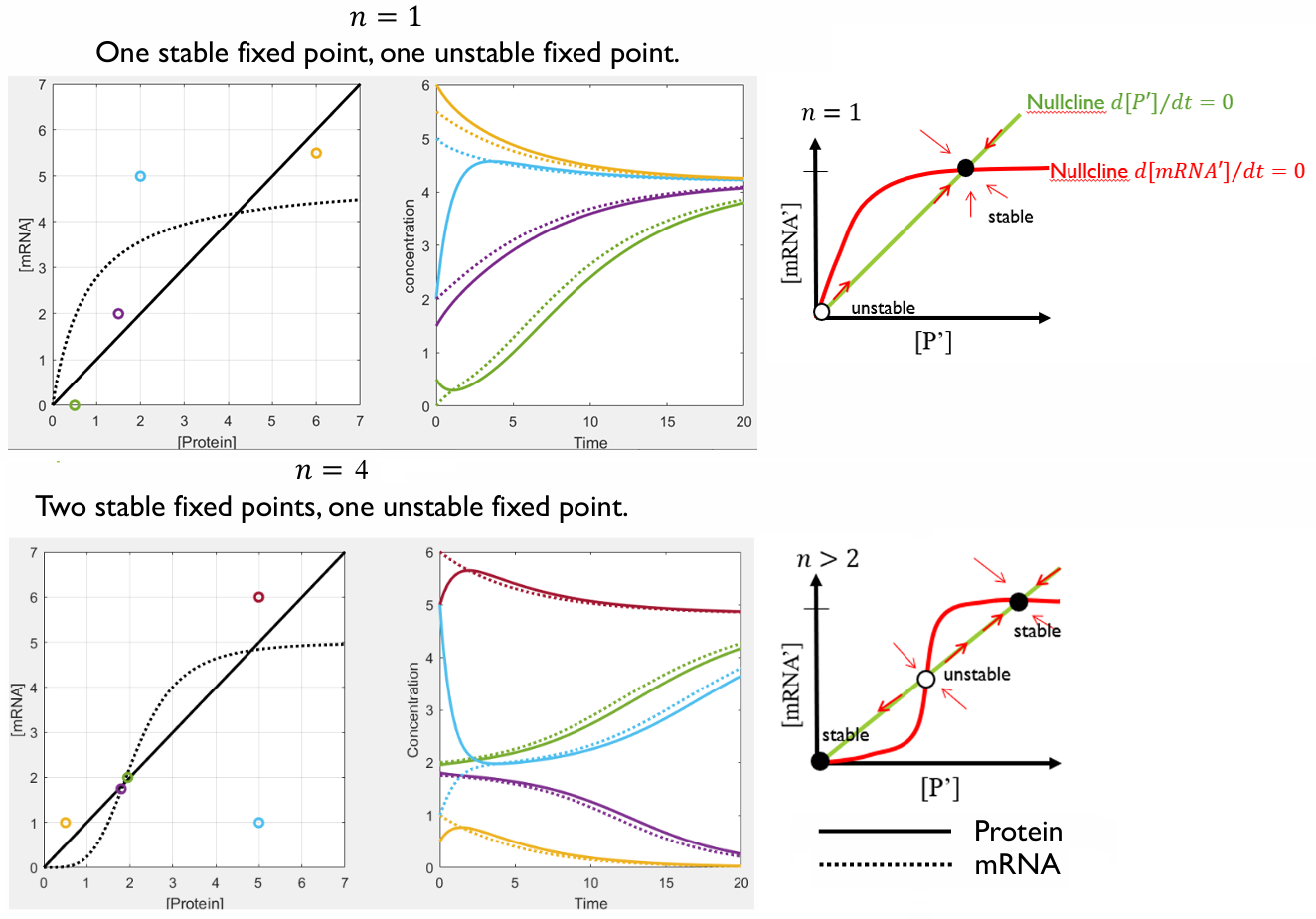
通过选点模拟,明显差异:
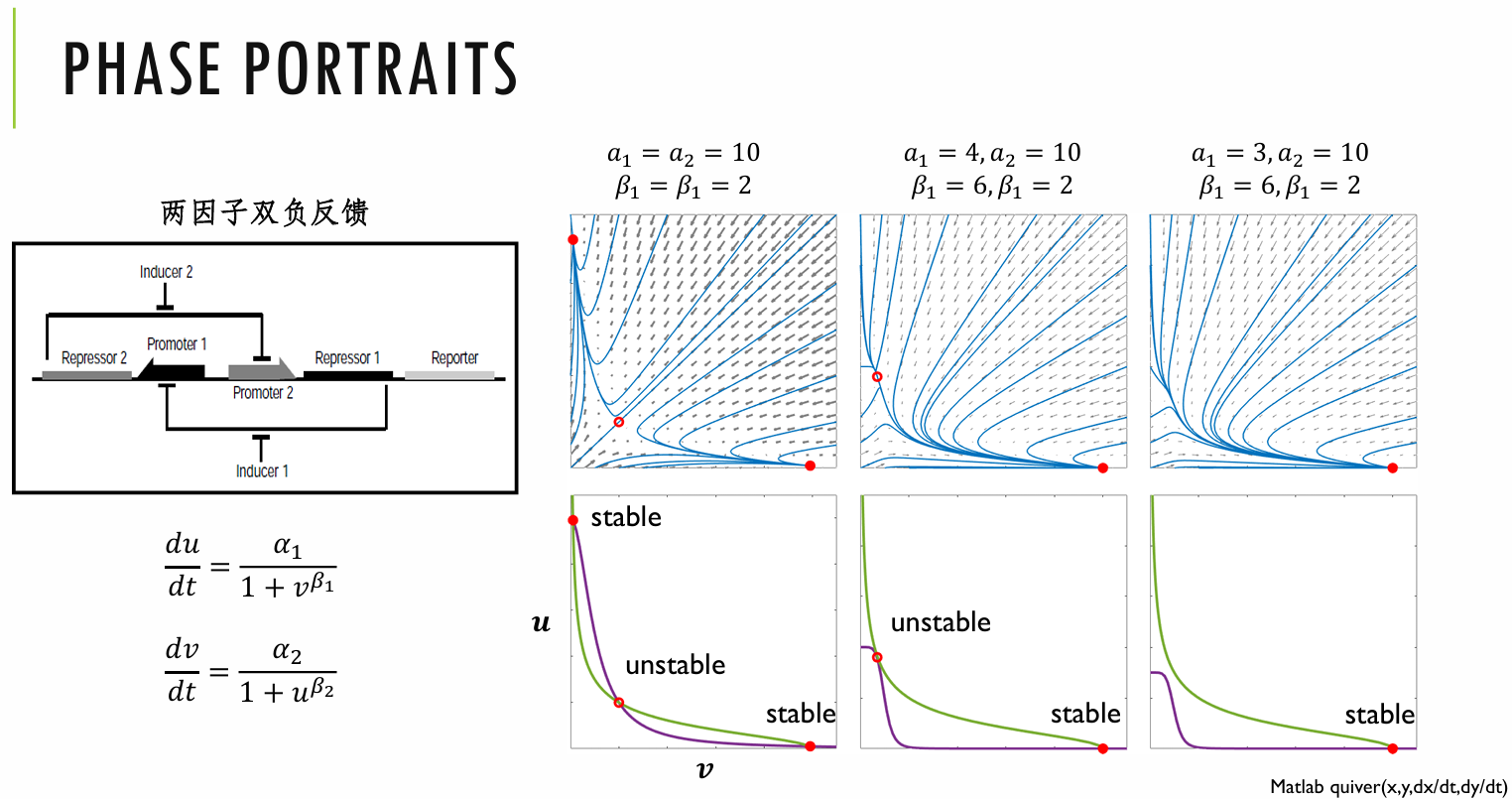
双因子负反馈,没有建模过程,主要是模拟,想看自戳
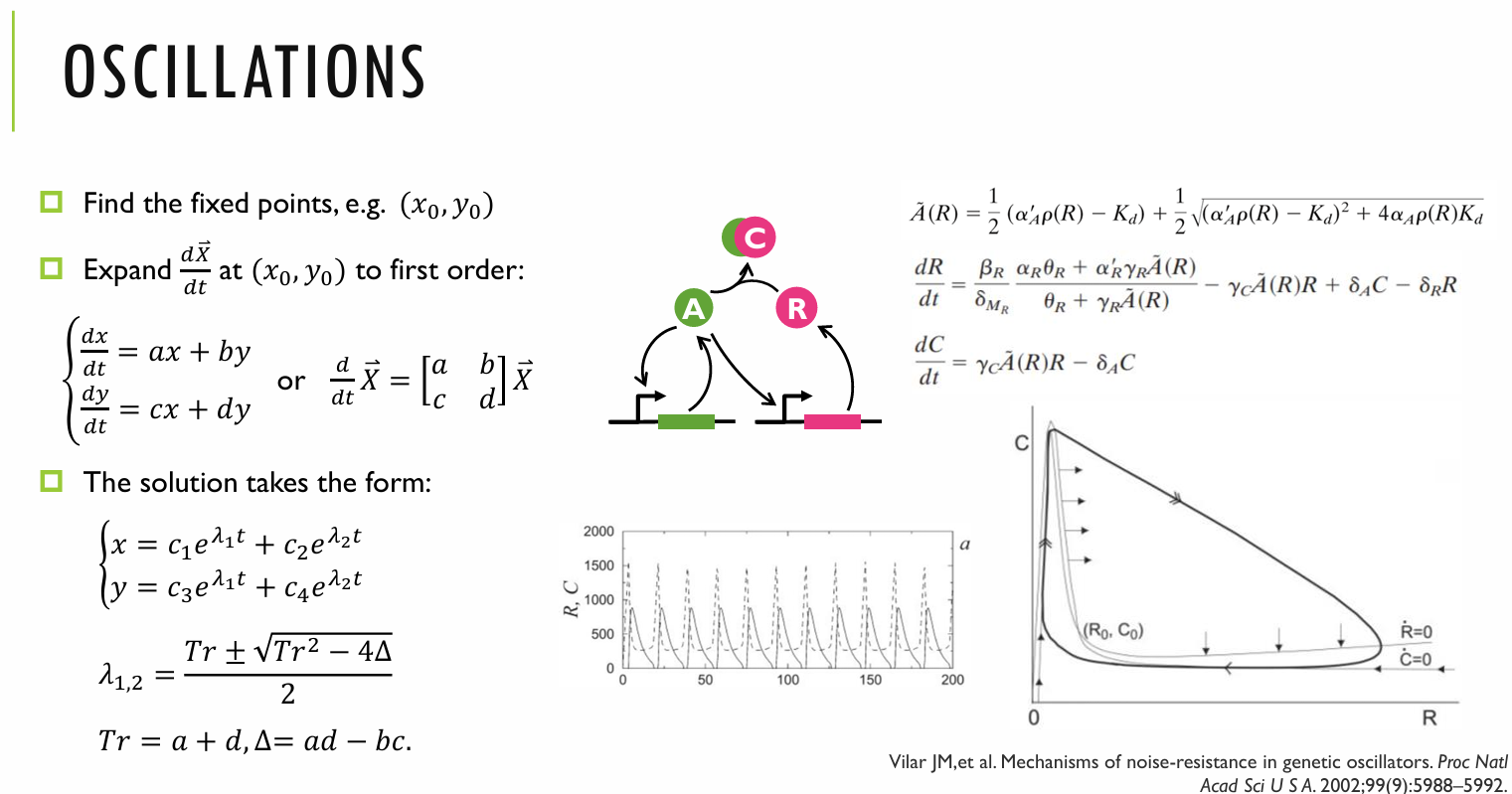
振荡(oscillations)建模,想看自戳
随机性,果蝇视觉细胞发育、感受态调控通路……
内噪声(引入后,转录调控等等,或者基因拷贝数增长差异)与外噪声(人工引入差异,比如基因拷贝数之类)。
随机模拟的gillespie algorithm:另外通过区分快慢反应可以简化模型。
Gillespie算法是一种用于模拟化学反应系统随机动力学行为的算法,特别适用于合成生物学中分子数量较少、反应随机性显著的系统(分子间碰撞偶然发生、反应发生与否受热力学涨落影响)。
Gillespie算法通过随机抽样来决定下一个反应事件及其发生时间。它基于马尔可夫过程,即系统的未来状态只依赖于当前状态。

8. 人工合成系统的模拟计算
Nothing Found Here.
9. 真核生物合成生物学
酵母合成生物学:two step gene replacement. 经历两次重组,分别是阳性筛选和阴性筛选,筛选标准是营养利用能力。
suspension cells、adhesion cells (贴壁,Hela).
Transient transfection (瞬时转染,可能会丢失)、稳定转染 (需要传多代)。如果转两至多个质粒,存在共转染现象。Lentivirus、Retrovirus. Transgene silencing (参与了宿主基因组的修饰,自身活性也被调节).
10. 基于合成生物学的疾病诊疗
工程化细菌回路、病毒载体、纳米载体、细胞疗法。基于RNAi的肿瘤特异性识别线路。
免疫细胞工程化思路。CAR、SynNotch。智能细胞(远红外定向诱导功能性神经细胞的分化)。
11.1 文献阅读与学术报告规范
明确目标→判断文章匹配度→背景图表粗略了解→结论部分深化了解→主体内容精读→补充材料勿忽视。
读人读典读新。
阅读标记工具、英文辅助阅读工具、笔记记录工具、内容升华工具(mindmap)。
11.2 实验报告
题目-实验者信息-目的-原理-材料-方法-结果-结论-讨论。
12. 代谢工程
现代代谢工程大致分为三类:系统生物学对代谢模型的定量描述与途径预测、合成生物学的工具调控代谢过程、定向进化工程改良代谢所需的酶促反应。
青蒿素、PHA可降解塑料。
13. 合成生物学伦理与安全
大豆玉米棉花油菜。合成生物学存在哪些安全或伦理道德问题,如何防范应对;如何看待基因改造的物种或生物产品;制定怎么样的个人原则与规范来避免违反安全及伦理问题。
推进基本知识和制造新产品;解决社会问题(粮食营养能源病害)。
合成微生物与环境或其他有机体可能产生始料不及的相互作用,从而对环境和公共卫生造成风险;合成微生物释放到环境中可能引起HGT并影响生态平衡,或发生演变产生异常功能,对环境和其他有机体产生前所未有的副作用。
- Title: iGEM
- Author: Kirschy
- Created at : 01-16-2025 00:00:00
- Updated at : 02-19-2025 09:58:32
- Link: https://kirschyr.github.io/2025/01/16/iGEM Notes/
- License: This work is licensed under CC BY-NC-SA 4.0.
