Norovirus

前言
诺如病毒(Norovirus),作为杯状病毒科(Calicivirus)中诺如病毒属各病毒的统称,是一种能在全球范围内引起人与多种动物发生急性肠胃炎(AGE)、导致严重腹泻的人畜共患病病原体。传播途径多样、感染剂量低、排毒时间长、环境抵抗能力强、变异速度快、免疫保护时间短……种种特点让诺如病毒具有相当高的传染性和快速传播能力,在秋冬季流行,时常出现局部爆发现象。
时至今日,都没有任何针对诺如病毒的疫苗或特效药物。尽管诺如病毒感染性腹泻是一种自限性疾病,但其传播仍然对人群健康造成严重威胁(尤其是对于幼儿和老年人等弱势群体来说,如果个体免疫功能低下还可能罹患导致营养不良的慢性腹泻,给患者带来更长期的痛苦)、并造成巨大的社会经济损失。据世界卫生组织估计,诺如病毒每年造成6.85亿个病例、其中2亿都是儿童病例,尽管诺如病毒致死率并不高,但在庞大基数下仍能造成200,000人死亡,其中约5万名都是儿童。
以粪口传播途径为主的诺如病毒在食品卫生领域可谓试金石,博主曾有幸在短短两周时间内从白鲸大雪食堂获取病原体两次(食的真是雪罢,悲),虽然没有呕吐但也严重腹泻,没有食欲昏昏沉沉地度过了总近四天的脱水时间,现在想来仍是心有余悸。不过也正是这样的经历才能让博主落笔于此吧。
侵袭
静观
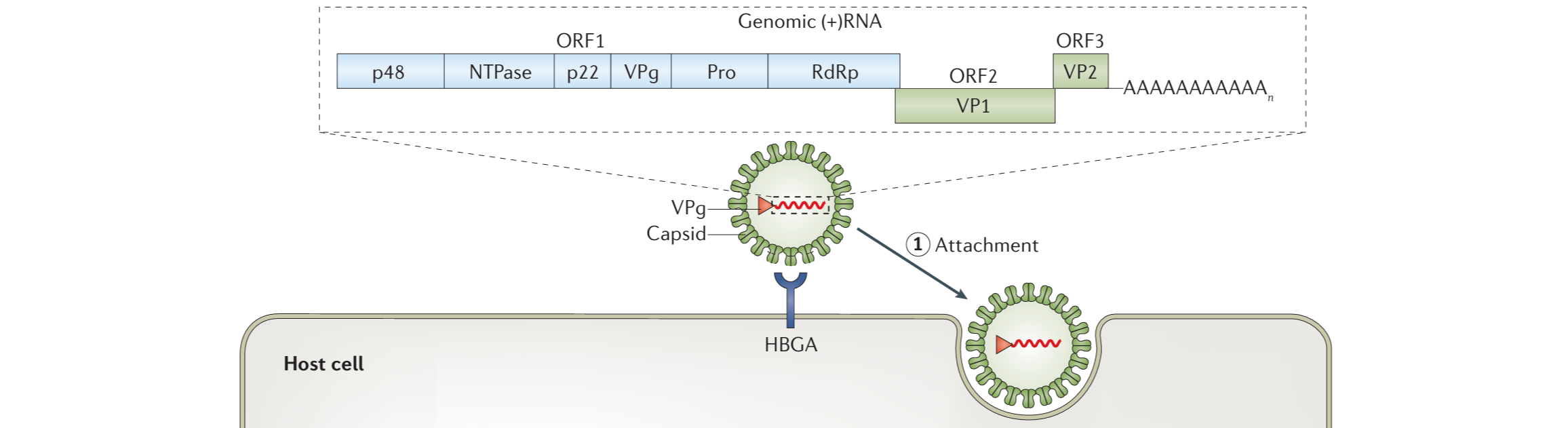
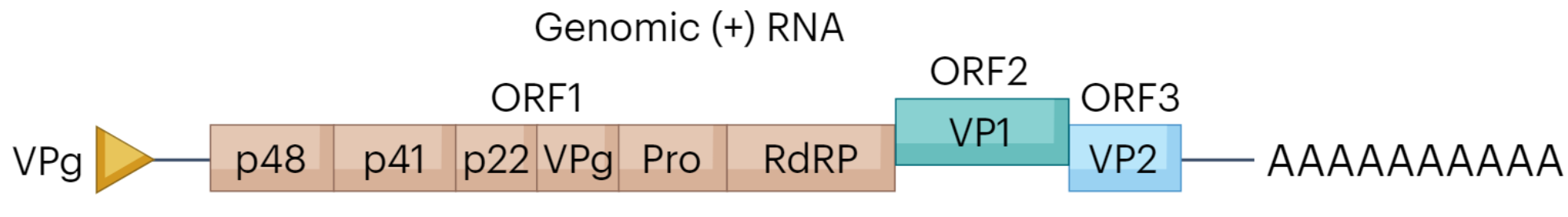
人类诺如病毒(HuNoV)基因组简单——仅有一条长约7.5kb的单链正义RNA。在+ssRNA的5’端具有一个共价连接的病毒蛋白Vpg,同时为了保证在真核细胞中的稳定性,基因组3’端与一般的mRNA一样具有polyA尾巴。基因组共有三个开放阅读框(ORF),多顺反子的ORF1所得聚合蛋白需要后续经病毒蛋白酶剪切才有功能,而另外ORF2和ORF3都为单顺反子。具体的病毒蛋白的功能在此列表不赘述。
| Type | Function |
|---|---|
| p48/NS1 or 2(ORF1) | |
| p41/NS3(ORF1) | 有解旋酶和RNA伴侣活性。诱导细胞内膜改变并与p22共同参与复制 |
| p22/NS4(ORF1) | 具有高度保守的内质网输出信号基序。破坏高尔基体,抑制细胞分泌 |
| Vpg/NS5(ORF1) | 作为复制时的引物;与帽结合eIF4G复合物互作 |
| 蛋白酶/NS6(ORF1) | 药物开发靶标。负责将ORF1各单体蛋白切开 |
| RdRP/NS7(ORF1) | 药物开发靶标 |
| 主要结构蛋白/VP1(ORF2) | 三重轴对称位 |
| 次要结构蛋白/VP2(ORF3) | 五重轴对称位,协助核酸+蛋白质壳二十面体组装 |
诺如病毒结构简单——无包膜的正二十面体蛋白质壳为其提供仅有的保护。VP1围绕二十面体的三重轴(3-fold axis)构建、VP2围绕二十面体的五重轴(5-fold axis)构建,三重轴相接处是为二重轴。在三重轴和五重轴相接的地方就是诺如病毒附着人类细胞表面分子HBGA的位点。
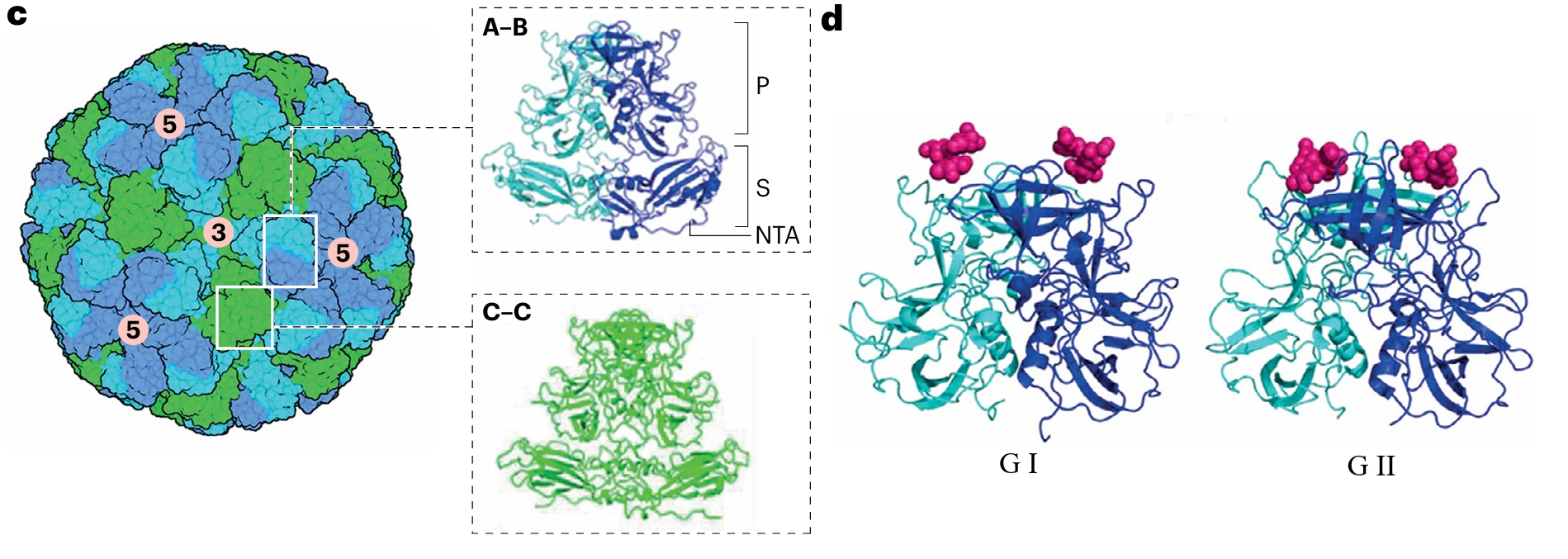
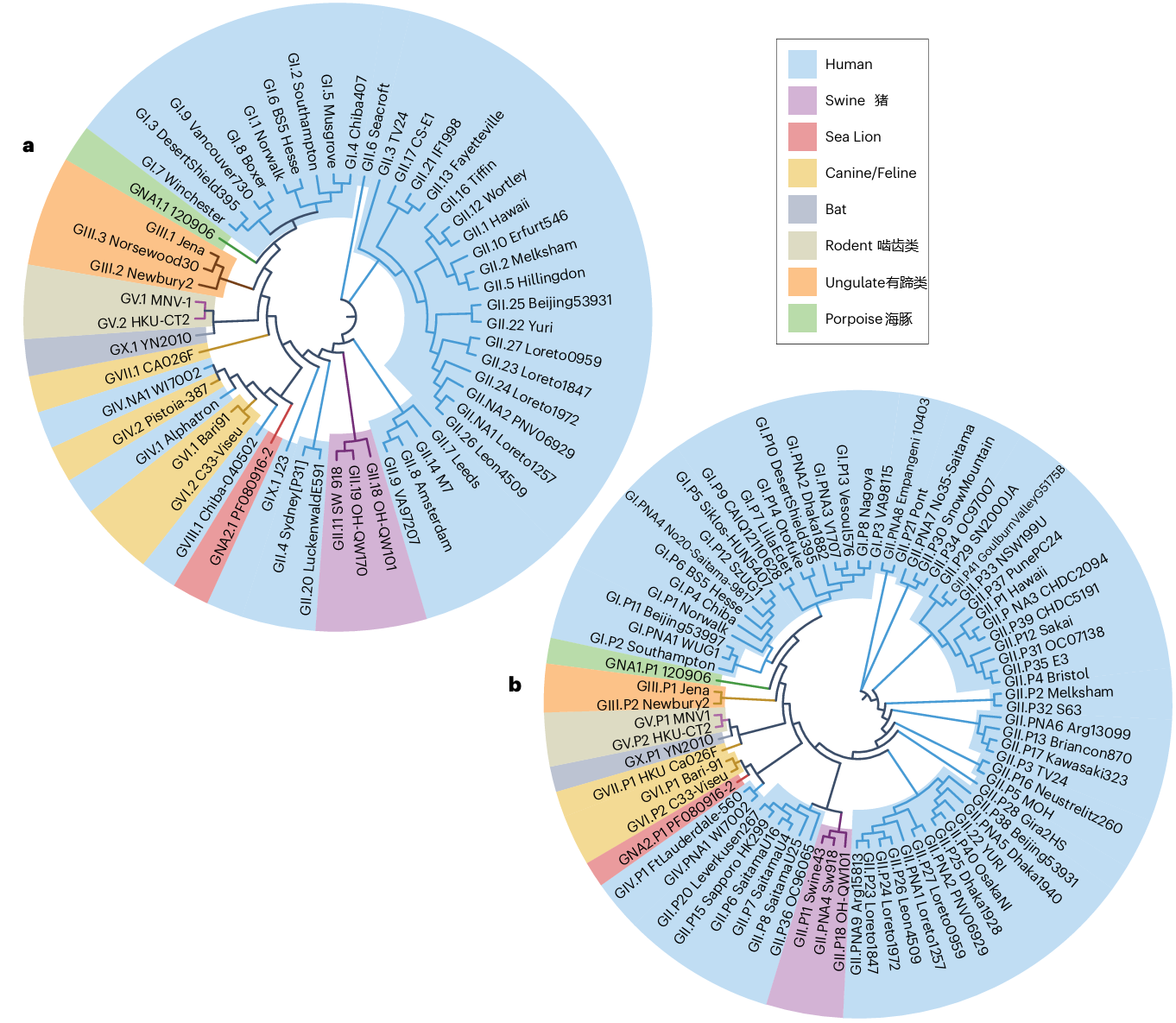
依据VP1的氨基酸序列进行系统分析,可以将诺如病毒属划分为遗传组群(genogroup)和更低一级的遗传型(genotype)层级。现在被认定的遗传组群共有10个(GI-GX)、遗传型共有48个。在GI、GII、GIV、GVIII、GIX几种能感染人类的遗传组群中,GII是在人群中最广为流传的一类,其中GII.4更是近二十年来的主流毒株。

其变
病毒的感染在细胞层次无非几个动作:附着(attachment)→内吞(internalization)→去包被去组装(uncoating and disassembly)→表达(translation+post-translational cleavage)→复制(replication)→组合包装(encapsidation)→释放(release)。
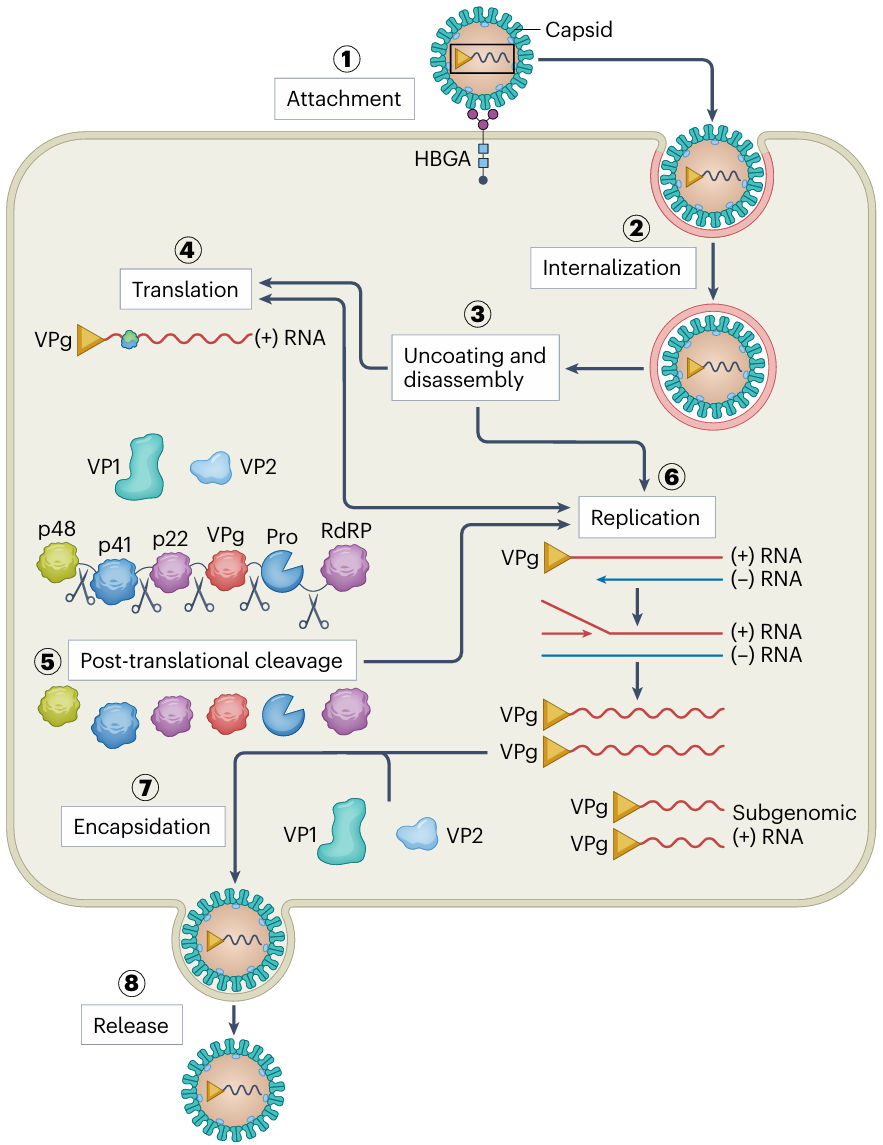
附着
诺如病毒的初始细胞受体是组织血型抗原(HBGA)聚糖,作为ABO血型的决定因素,在分泌物、肠道上皮细胞中广泛存在。也因此人群对于诺如病毒属于普遍易感,少数被发现对绝大多数诺如病毒毒株都表现出抗性的个体一般都具有FUT2突变。FUT2表达产物为半乳糖苷 α-(1,2)-岩藻糖基转移酶2,而实际上观察HBGA的结构就很容易发现背后的关联。
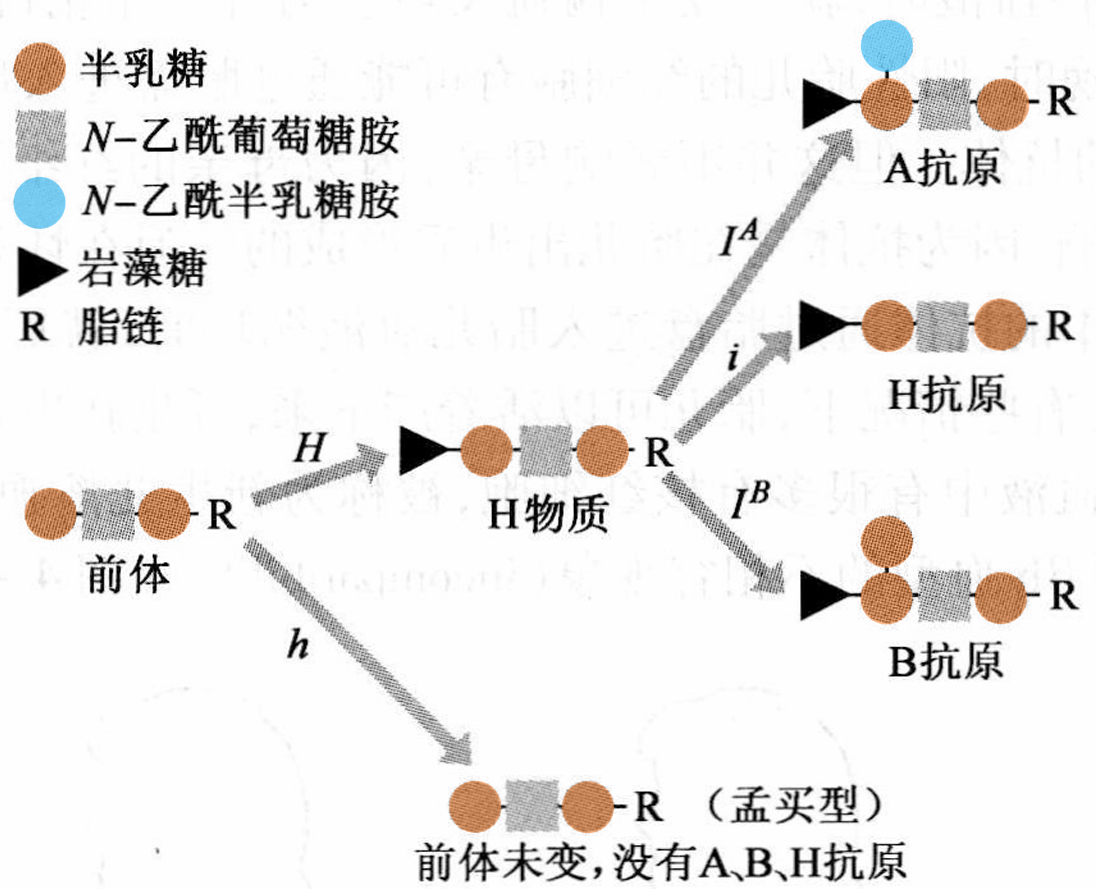
FUT2的直接功能是催化L-岩藻糖从鸟苷二磷酸-β-L-岩藻糖转移到细胞表面糖蛋白和糖脂的O-和N-连接聚糖链上的末端半乳糖上,它实际上也就是一众遗传学教材中会提到的“H基因”的真身(的孪生兄弟)。既然这里是FUT2,那么自然就会有FUT1,二者在催化功能方面没有太大差异,主要是在表达位置上,FUT1主要在中胚层来源的细胞中表达、FUT2主要在内胚层来源的细胞中表达。也因此,FUT2通过介导糖蛋白糖脂的H抗原形成,影响宿主与肠道微生物的互作。
因此,这里有一个有趣的事实是:孟买血型的人极大概率都不会被诺如病毒感染。而为什么说“极大概率”——因为仍然有GII.3的部分株系和极罕见极偶然的GII.4感染例外。推测是个体的微生物种群特殊,微生物给肠上皮细胞的H抗原前体加上了半乳糖变成了可供附着感染的H抗原。
内吞与逃逸
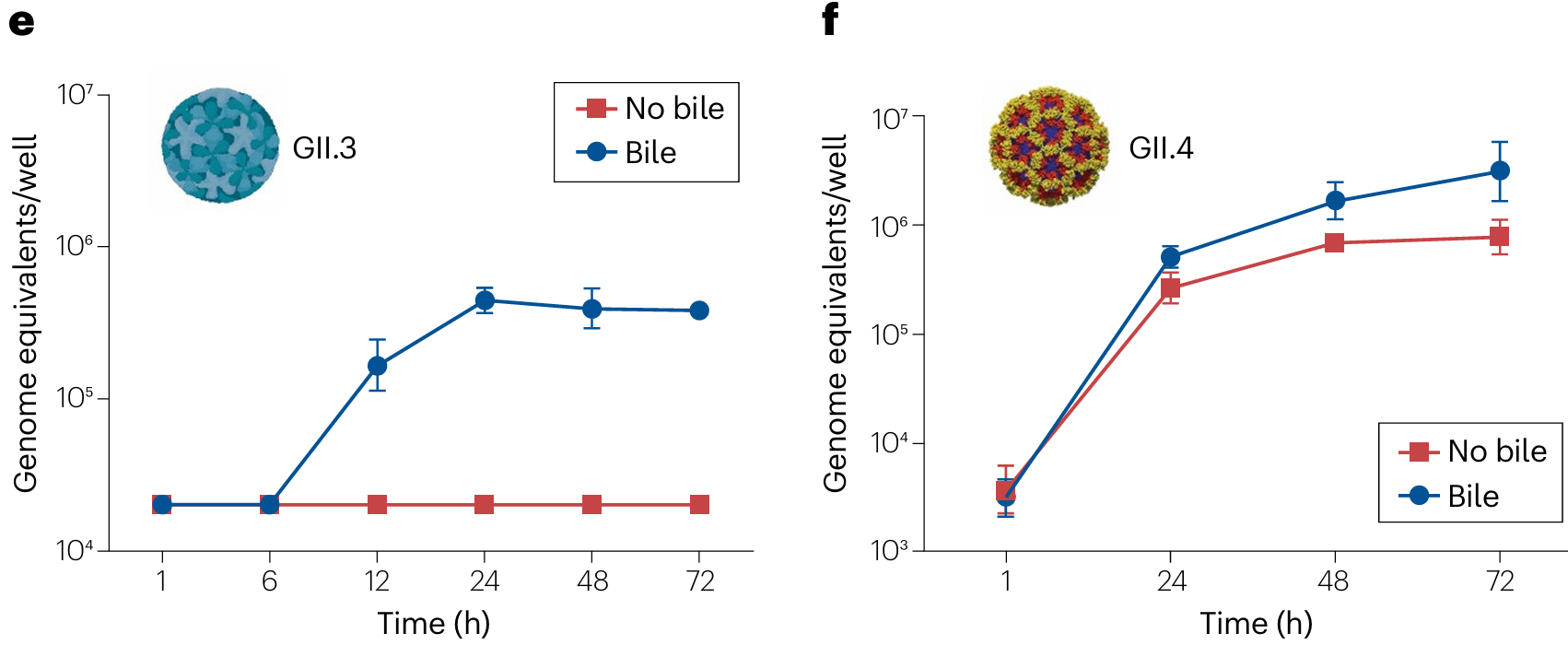
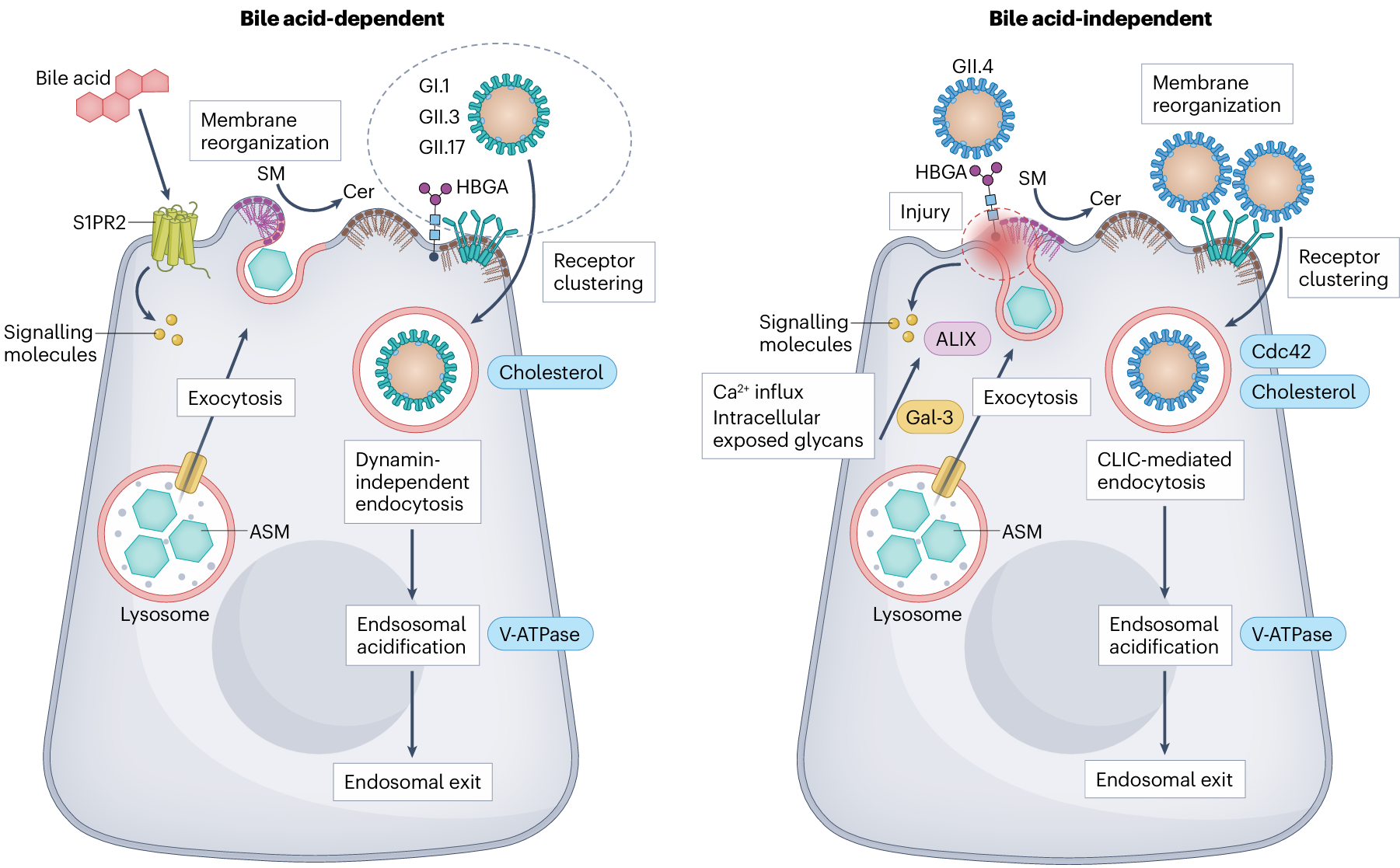
当诺如病毒与HBGA结合后,通过内吞进入细胞中,然而这个过程即便在同一个遗传组群中都有所不同——比如GII.3需要胆汁而GII.4不需要,但是胆汁能增强GII.4的复制,而除了GII.3,另外在GII下还有1、6、17等基因型有胆汁依赖性。
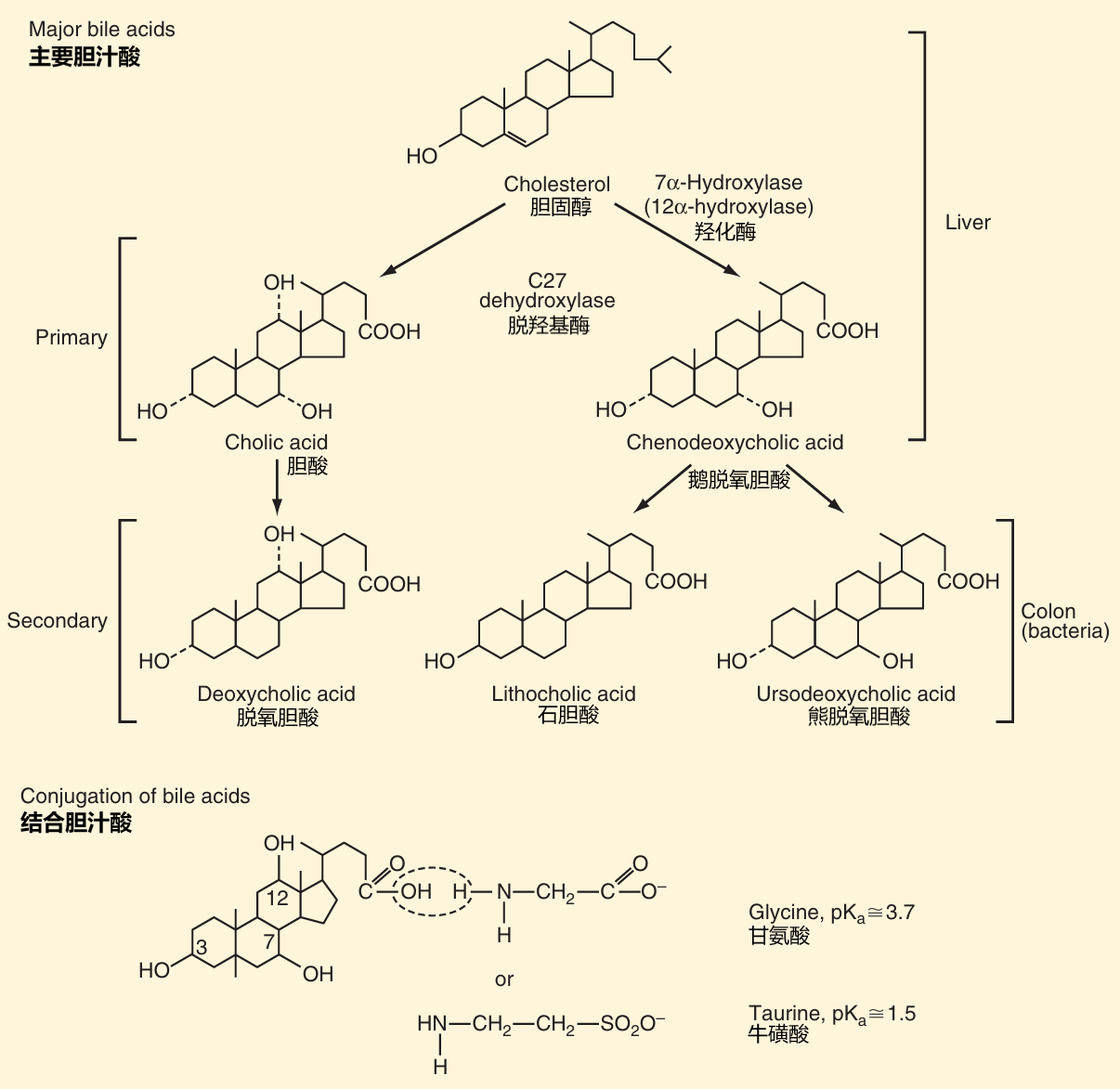
然而胆汁成分复杂,含无机盐、胆汁酸、胆色素、胆固醇、卵磷脂和黏蛋白等等,而其中有效成分到底是什么,怎么协助这些胆汁依赖性的毒株进行感染,都是近年来才明确的问题。对不同种哺乳动物胆汁进行热处理和胰蛋白酶处理后,其仍然具有协助GII.3侵染并复制的能力、GII.4的复制活性也依然有一定程度加强,说明协助侵染的是一种高度保守的非蛋白组分。通过对胆汁中主要有效成分进行测试,发现胆汁酸(bile acid)对GII.3侵染有主要作用。
胆汁酸同样具有多种类型,十分多样,包含胆酸(CA)、鹅脱氧胆酸(CDCA)等初级胆汁酸,还有经结肠细菌代谢形成的次级胆汁酸,胆汁酸与甘氨酸或牛磺酸连接形成胆盐(bile salt),在消化过程中承担乳化功能,也避免高疏水性胆汁酸对肠道上皮细胞的质膜造成损伤。
在模拟的空肠环境中用各种不同的胆汁酸来测试GII.3的复制能力,发现基因组扩增程度与所使用的胆汁酸之疏水性显著相关。根据实验结果以及出于对高浓度强疏水性胆汁酸细胞毒性和溶解性好的考虑,针对具有强辅助GII.3复制作用的甘氨酸鹅脱氧胆酸(GCDCA)来进一步开展研究。通过在不同的时间点添加GCDCA,可以确定GCDCA的作用时间,GCDCA在感染实现前3小时添加能实现最佳效果,说明GCDCA促进病毒进入细胞,然后进行基因组脱壳、非结构蛋白表达和基因组复制,从而产生产生VP1组装后代病毒所需的基因组RNA。
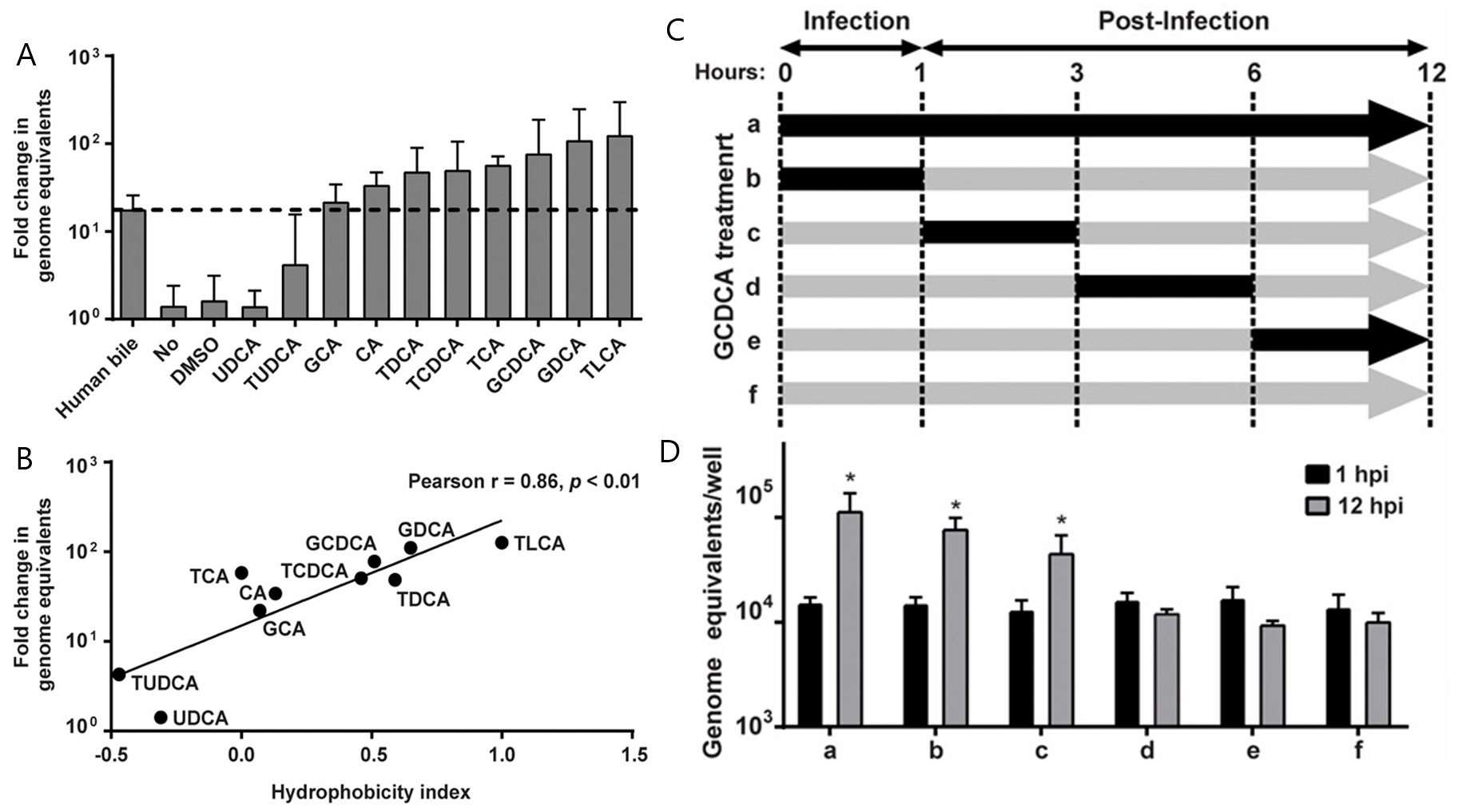
事实上,胆汁酸的正常生理功能也并非从来只有乳化促进消化(“洗涤剂效应”)这一条,各种胆汁酸还可以激活GPCR、法尼醇X核受体(FXR)、Takeda G蛋白偶联受体5(TGR5)等来产生其他生理影响。通过使用其他去污剂、GPCR/FXG/TGR5的激活剂或抑制剂处理,都未发现GII.3的感染复制有受到影响,否定了以上的可能性(但高通量RNA测序发现FXG下游靶标转录上调,原因未阐明)。大规模筛选胆汁酸的可能受体并施用抑制剂后,最终确定GCDCA等胆汁酸通过鞘氨醇-1-磷酸受体2(S1PR2)协助GII.3进行感染。
S1PR2:也是一种GPCR。虽名鞘氨醇-1-磷酸受体,但其配体并不仅限于鞘氨醇-1-磷酸。另外还有其他脂质(甘油三酯、类固醇、甘油二酯、糖脂、磷脂、脂肪酸……)、整合素(参与细胞-基质粘附)也都可以与之结合。
当GII.3被内吞后,其所在的内体将会酸化,这种酸化是依赖于胆汁酸处理的。内体酸化作为绝大多数经内吞途径侵染的病毒进入细胞质的关键,这类病毒(埃博拉病毒、呼肠孤病毒和其他杯状病毒)大多会被组织蛋白酶切割而改变结构,能够从内体/溶酶体中逃逸,然而使用各种组织蛋白酶抑制剂都没能影响到GII.3的复制。那么就只能考虑溶酶体中其他酶的可能作用——酸性鞘磷脂酶(ASM)——被筛选出来。ASM在内体/溶酶体中负责将鞘磷脂分解为神经酰胺(Cer)。
最后一张图比较胆汁依赖和胆汁非依赖的诺如病毒在内吞侵染过程中的差异。
表达
内吞后便是去包被去组装,为在细胞质基质中表达与复制做好准备。作为单链正义RNA病毒,其RNA结构与行为与真核生物mRNA十分相似。VPg以一个大蛋白的形式作为“帽子”,在与帽结合eIF4G复合物互作的同时也能当RdRP复制RNA时的引物。
破坏
p41/NS3与p22/NS4一起能够通过caspase8和caspase9依赖性途径诱导细胞凋亡。
肆虐
追索
困局
RdRP的低保真率让诺如病毒的基因组突变率相当高。衣壳蛋白的突变也可能增加识别的广度和对HBGA的更强结合亲和力,如新出现的GII.17和GII.4菌株所述。
早在1972年,人类就在电子显微镜下首次观察到了诺如病毒,然而53年过去,依然没有任何针对诺如病毒的特效药或诺如病毒疫苗问世,它们始终在世界各个地方流窜、间断性地造成局部的疫情爆发,然后又迅速销声匿迹。而对于下一次的爆发,人们只能呼吁食品安全、关注对症下药,或者更多地独自煎熬。造成这种尴尬困局的核心原因是难以使用常规组织培养细胞和人类胎儿器官培养物培养病毒,且难以选择其他合适的动物模型来培养诺如病毒,对于一些可以感染诺如病毒动物模型,也只能实现体内感染,且这些动物模型昂贵而难以开展实验。
尽管如此,一些能够支持诺如病毒复制的动物模型依然在研究当中。例如敲除重组激活基因1或2(Rag)和共同γ链的BALB/c小鼠腹膜注射培养、斑马鱼幼体的卵黄囊注射培养等等,都主要关注GII.4模型的构建。而一些非人灵长类的研究则主要集中于猕猴,探究接种方式与易感性、病毒在粪便中的时空存在序列等方面。
少数细胞系在转染诺如病毒RNA后可以产生低水平病毒颗粒,尽管具有传染性,但仅会进行一轮RNA复制,再起不能。
重构
从历来的失败经验看来,培养诺如病毒需要构建一个与原生肠道系统极其相似的系统,因为单独的细胞培养中诺如病毒的繁衍难以延续可以说是“咄咄怪事”,势必存在其他影响诺如病毒感染与存活的因素,例如免疫细胞、肠道细菌等等。
有一些零散且重复难度较大的证据表明,特殊细菌(阴沟肠杆菌-Enterobacter cloacae)胞表的HBGA帮助了HuNoV阳性的粪便滤液恢复感染性。HuNoV可以在原代和传代B细胞中复制,但复制程度弱(复制10~50倍),一些B细胞缺陷患者更能大量排出毒粒。
从历次失败的培养尝试来看,HuNoV在体外的复制取决于模拟肠上皮细胞分化阶段的能力,也可能取决于细胞培养系统的微环境。在培养需求不甚明晰的情况下,人肠源类肠器官(human intestinal enteroid,HIE) 或 人组织来源类器官(tissue-derived organoids)成为了一种稳定且重复性好的培养系统选择。进行一定程度的分化后,HIE模型与人类小肠上皮在结构、生理、微环境等方面十分相似,甚至表现出昼夜节律,HuNoV亦实现了成功感染。
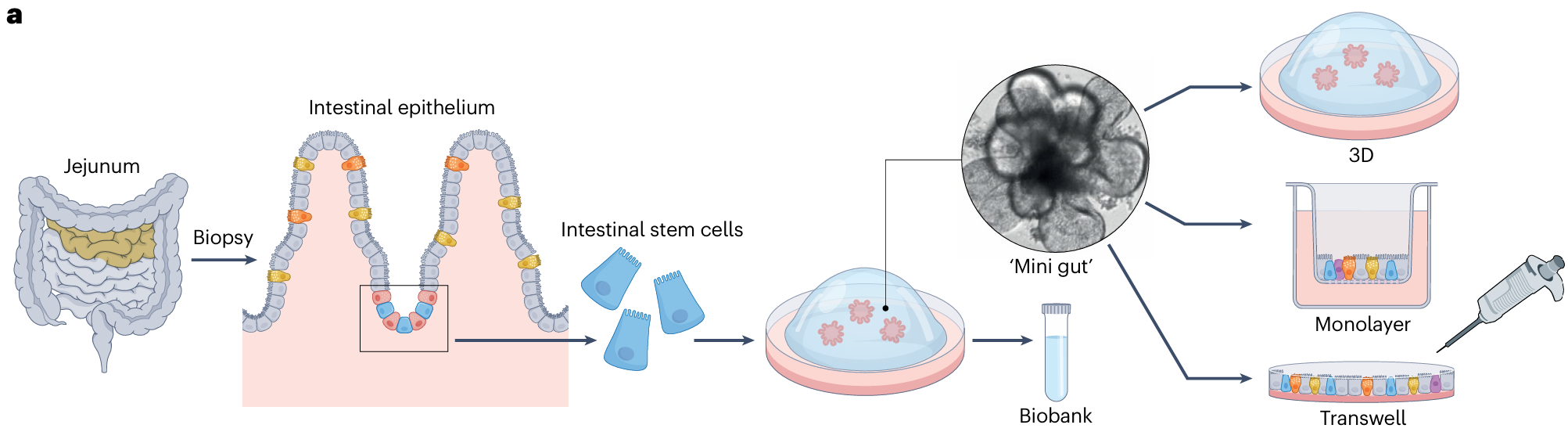
HIE的构建:来源于含肠道干细胞的肠组织样本,在含生长因子的培养基中自组织成3D结构,之后再去除生长因子,干细胞就会分化为成熟肠上皮中所具有的多种细胞类型。形成的培养物又可以转入其他多种形式的培养器皿中进行研究。
类器官中提供了对真实环境最接近的模拟(除了很多乱七八糟的肠道细菌之外),对HuNoV繁殖需求的研究得到满足(在前文提到的如胆汁依赖性,也是在类器官模型发展起来后才取得的成果),也明确了诺如病毒对小肠的独特嗜性(对大肠部分的结肠无感)。
奇特的一点是,受感染的类器官培养物中,只有吸收细胞(肠上皮细胞)被感染,而分泌细胞(肠内分泌细胞+杯状细胞+潘氏细胞)则无虞,与实际情况存在冲突——免疫低下个体的分泌细胞中有HuNoV复制,到底是HIE中分泌细胞少还是缺乏某种可能发生潜在感染的分泌细胞仍然不得而知。
另外还有由胚胎干细胞(多潜能干细胞)或多能干细胞分化而来的人肠道类器官(human intestinal organoid,HIO)同样支持一些诺如病毒毒株的生长复制。但HIO模型中细胞的分化水平相对更低,其肠上皮细胞对感染的容忍程度更低,难以维持高滴度载量 。
破局
从传统的抗病毒药物制造观点来看,针对诺如病毒复制过程的主要就是蛋白酶抑制剂和RdRP抑制剂。尽管目前已经发现对二者的一些小分子抑制剂,但是实际效果非常局限,在不同遗传组群之间效果差异大,而根本原因就是诺如病毒突变率高。
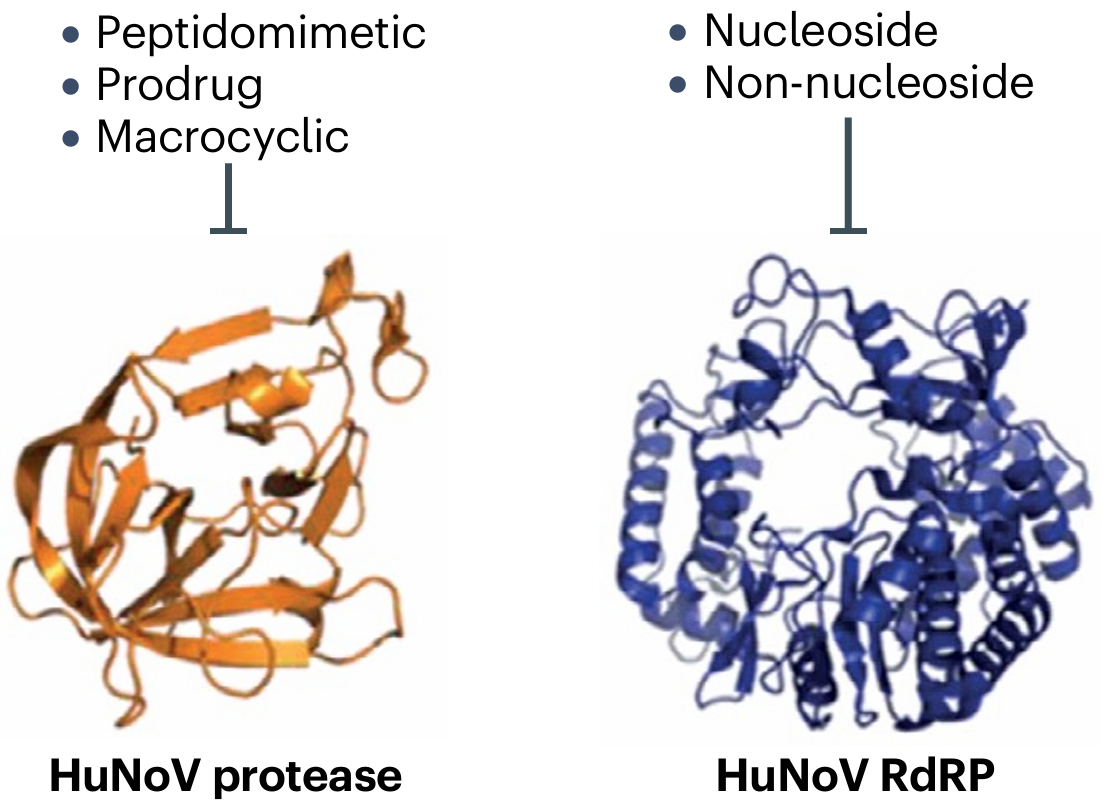
在普遍的遗传群组差异的背景下,需要找到一个诺如病毒属几乎所有成员都会表现出的特征,以此为靶标设计更有效的药物成为了更现实的难题。近来有研究发现诺如病毒RdRP在感染过程中会诱导液-液相分离,以相分离凝聚物作为复制枢纽,这为开发新的抗病毒药物开辟了路径。
References:
- Prasad BVV, Atmar RL, Ramani S, Palzkill T, Song Y, Crawford SE, Estes MK. Norovirus replication, host interactions and vaccine advances. Nat Rev Microbiol. 2025 Jan 17. doi: 10.1038/s41579-024-01144-9. Epub ahead of print. PMID: 39824927.
- Kaundal S, Anish R, Ayyar BV, Shanker S, Kaur G, Crawford SE, Pollet J, Stossi F, Estes MK, Prasad BVV. RNA-dependent RNA polymerase of predominant human norovirus forms liquid-liquid phase condensates as viral replication factories. Sci Adv. 2024 Dec 20;10(51):eadp9333. doi: 10.1126/sciadv.adp9333. Epub 2024 Dec 20. PMID: 39705355; PMCID: PMC11661447.
- Carlson KB, Dilley A, O’Grady T, Johnson JA, Lopman B, Viscidi E. A narrative review of norovirus epidemiology, biology, and challenges to vaccine development. NPJ Vaccines. 2024 May 29;9(1):94. doi: 10.1038/s41541-024-00884-2. PMID: 38811605; PMCID: PMC11137017.
- Wang G, Zhang D, Orchard RC, Hancks DC, Reese TA. Norovirus MLKL-like protein initiates cell death to induce viral egress. Nature. 2023 Apr;616(7955):152-158. doi: 10.1038/s41586-023-05851-w. Epub 2023 Mar 29. PMID: 36991121; PMCID: PMC10348409.
- Winder N, Gohar S, Muthana M. Norovirus: An Overview of Virology and Preventative Measures. Viruses. 2022 Dec 16;14(12):2811. doi: 10.3390/v14122811. PMID: 36560815; PMCID: PMC9781483.
- Ford-Siltz LA, Tohma K, Parra GI. Understanding the relationship between norovirus diversity and immunity. Gut Microbes. 2021 Jan-Dec;13(1):1-13. doi: 10.1080/19490976.2021.1900994. PMID: 33783322; PMCID: PMC8018473.
- Ludwig-Begall LF, Mauroy A, Thiry E. Noroviruses-The State of the Art, Nearly Fifty Years after Their Initial Discovery. Viruses. 2021 Aug 4;13(8):1541. doi: 10.3390/v13081541. PMID: 34452406; PMCID: PMC8402810.
- Campillay-Véliz CP, Carvajal JJ, Avellaneda AM, Escobar D, Covián C, Kalergis AM, Lay MK. Human Norovirus Proteins: Implications in the Replicative Cycle, Pathogenesis, and the Host Immune Response. Front Immunol. 2020 Jun 16;11:961. doi: 10.3389/fimmu.2020.00961. PMID: 32612600; PMCID: PMC7308418.
- Murakami K, Tenge VR, Karandikar UC, Lin SC, Ramani S, Ettayebi K, Crawford SE, Zeng XL, Neill FH, Ayyar BV, Katayama K, Graham DY, Bieberich E, Atmar RL, Estes MK. Bile acids and ceramide overcome the entry restriction for GII.3 human norovirus replication in human intestinal enteroids. Proc Natl Acad Sci U S A. 2020 Jan 21;117(3):1700-1710. doi: 10.1073/pnas.1910138117. Epub 2020 Jan 2. Erratum in: Proc Natl Acad Sci U S A. 2020 Jul 7;117(27):16084. doi: 10.1073/pnas.2011295117. PMID: 31896578; PMCID: PMC6983410.
- Green KY, Kaufman SS, Nagata BM, Chaimongkol N, Kim DY, Levenson EA, Tin CM, Yardley AB, Johnson JA, Barletta ABF, Khan KM, Yazigi NA, Subramanian S, Moturi SR, Fishbein TM, Moore IN, Sosnovtsev SV. Human norovirus targets enteroendocrine epithelial cells in the small intestine. Nat Commun. 2020 Jun 2;11(1):2759. doi: 10.1038/s41467-020-16491-3. PMID: 32488028; PMCID: PMC7265440.
- Hassan E, Baldridge MT. Norovirus encounters in the gut: multifaceted interactions and disease outcomes. Mucosal Immunol. 2019 Nov;12(6):1259-1267. doi: 10.1038/s41385-019-0199-4. Epub 2019 Sep 9. PMID: 31501514; PMCID: PMC7479810.
- Netzler NE, Enosi Tuipulotu D, White PA. Norovirus antivirals: Where are we now? Med Res Rev. 2019 May;39(3):860-886. doi: 10.1002/med.21545. Epub 2018 Dec 25. PMID: 30584800; PMCID: PMC7168425.
- de Graaf M, van Beek J, Koopmans MP. Human norovirus transmission and evolution in a changing world. Nat Rev Microbiol. 2016 Jul;14(7):421-33. doi: 10.1038/nrmicro.2016.48. Epub 2016 May 23. PMID: 27211790.
- FUT2 - Galactoside alpha-(1,2)-fucosyltransferase 2 - Homo sapiens (Human) | UniProtKB | UniProt
- S1PR2 - Sphingosine 1-phosphate receptor 2 - Homo sapiens (Human) | UniProtKB | UniProt
- WHO-Norovirus-Immunization, Vaccines and Biologicals
- Jane Flint. Principles of Virology (Fifth Edition) [M]. Washington, DC: ASM press, WILEY, 2020.
- Bruce M. Koeppen, Bruce A. Stanton. Berne & Levy Physiology (Seventh Edition) [M]. Philadelphia, PA: Elsevier, 2018.
- 刘祖洞. 遗传学 (第三版) [M]. 北京: 高等教育出版社, 2013.
- 王庭槐. 生理学 (第三版) [M]. 北京: 人民卫生出版社, 2015.
Recommendations:
- Collins SL, Stine JG, Bisanz JE, Okafor CD, Patterson AD. Bile acids and the gut microbiota: metabolic interactions and impacts on disease. Nat Rev Microbiol. 2023 Apr;21(4):236-247. doi: 10.1038/s41579-022-00805-x. Epub 2022 Oct 17. PMID: 36253479.
Ongoing update……
- Title: Norovirus
- Author: Kirschy
- Created at : 01-20-2025 00:00:00
- Updated at : 02-09-2025 11:28:43
- Link: https://kirschyr.github.io/2025/01/20/Norovirus/
- License: This work is licensed under CC BY-NC-SA 4.0.
